使用python做图像处理
安装python程序包mahotas
如果是windows下使用pythonxy,可以在这里下载mahotas作为插件。如果是linux或者ios,可以直接easy install。
对生物细胞图像的分析
原文连接见这里
热身
先调用一些常用包
import numpy as np
import scipy
import matplotlib.pyplot as plt
import mahotas
from scipy import ndimage
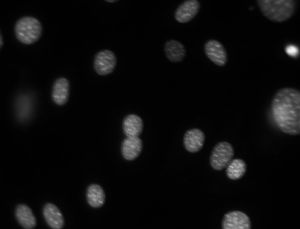
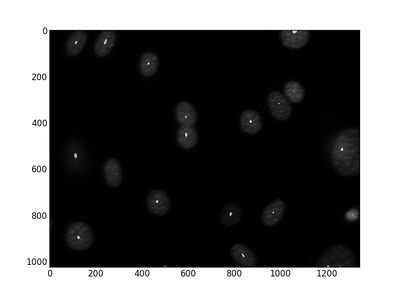
读入一张图。如下所示(实验代码时可以从本网页下载并保存这张图)。
dna = mahotas.imread('E:/wulingfei/image_processing/dna_raw.jpeg')
#dna.shape
plt.gray()
plt.imshow(dna)
#dna.max()
#dna.min()
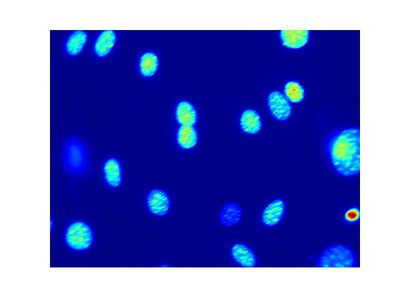
dna_raw.jpeg被读进来后,变成一个1024行,1344列的numpy array。所以用plt.imshow()可以直接画出来。plt.imshow有两种上色机制,plt.gray()和plt.jet(),后者是默认机制。因为我们这里设定plt.gray()所以画出来是黑白的。如果没有设定的话,画出来就是彩色的(通过plt.axis('off')移除坐标轴):
plt.jet()
plt.imshow(dna)
plt.axis('off')
plt.savefig('E:/wulingfei/image_processing/dna_1.jpeg', bbox_inches=None)
计算细胞的数量
为了计算细胞的数量,我们要先对图像进行一些处理。首先,我们要设定一个阈值后,把图像变成黑白的。
plt.gray()
T = mahotas.thresholding.otsu(dna)
plt.imshow(dna > T)
plt.savefig('E:/wulingfei/image_processing/dna_2.jpeg', bbox_inches=None)
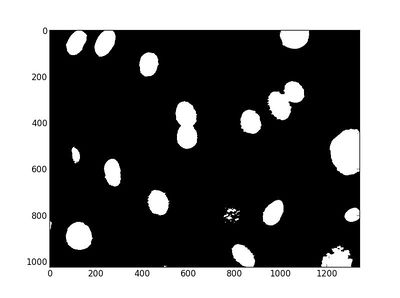
转换成黑白图像后,我们就可以通过计算连接在一起的components的个数来得到细胞的数量。不过现在的图像太粗糙,会大大影响我们对联系在一起的component的计算,所以我们先加上高斯过滤让它的边缘变得光滑
dnaf = ndimage.gaussian_filter(dna, 8)
T = mahotas.thresholding.otsu(dnaf)
plt.imshow(dnaf > T)
plt.savefig('E:/wulingfei/image_processing/dna_3.jpeg', bbox_inches=None)
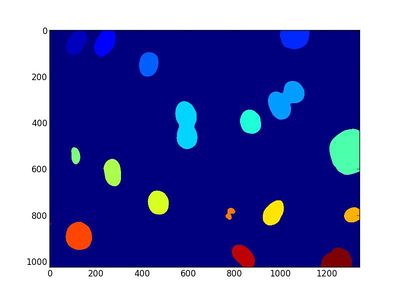
现在,我们可以在矩阵中划分团块并且给出不同团块的数量了。一共有18个团块,我们给它打上标签并且以不同的颜色表示。
labeled,nr_objects = ndimage.label(dnaf > T)
nr_objects
plt.imshow(labeled)
plt.jet()
plt.savefig('E:/wulingfei/image_processing/dna_4.jpeg', bbox_inches=None)
任务完成!
更精确地计算细胞的数量
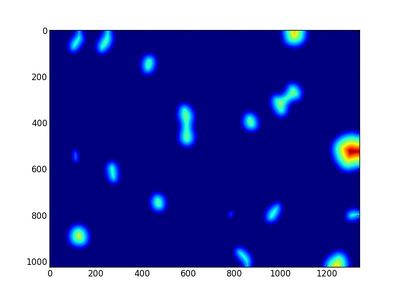
但是且慢,有一些细胞在照片上重叠了,被我们算作一个细胞了。显然是这不科学的,因此我们需要更精确的计算方法。接下来我们要讨论的方法寻找团块的中心点并计算中心点的个数。这里我们假设在灰度图上,团块比较中心的地方比较亮,最亮的地方就是最中心的地方。这个东西叫regional maxima,相当于一片山脉中的最高峰。我们找到这个点之后,进行标亮,并且与原来的灰度图重叠在一起。
dnaf = ndimage.gaussian_filter(dna, 16)
rmax = mahotas.regmax(dnaf)
plt.gray()
plt.imshow(dna)
plt.imshow(rmax,alpha=0.6)
seeds,nr_nuclei = ndimage.label(rmax)
plt.savefig('E:/wulingfei/image_processing/dna_5.jpeg', bbox_inches=None)
正如nr_nuclei所告诉我们的那样,一共有22个细胞。
划分细胞的活动区域
T = mahotas.thresholding.otsu(dnaf)
dist = ndimage.distance_transform_edt(dnaf > T)
plt.jet()
plt.imshow(dist)
对于矩阵中不为0的任意一个点i,ndimage.distance_transform_edt会计算与之最接近的背景点j(矩阵中等于0的点)与i的欧式距离。这意味着,越大的团块的中心点数值越大,表达成热图的话,颜色越浓:
当然,我们也可以把颜色反转(使用矩阵最大值减去矩阵内所有元素,使得矩阵元素的数值原来大的变小,小的变大),得到这个图:
dist = dist.max() - dist
dist -= dist.min()
dist = dist/float(dist.ptp()) * 255
dist = dist.astype(np.uint8)
plt.jet()
plt.imshow(dist)
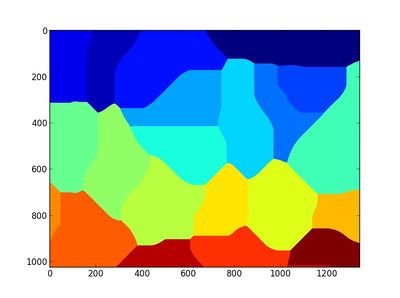
现在,根据矩阵的元素的大小,以之前得到的一堆最高峰(seeds)为核心位置,观察每个核心与邻居核心之间的边界(元素数值的局域极大值),把这个边界标记出来。就得到了核心的区域划分图。
nuclei = mahotas.cwatershed(dist, seeds)
plt.jet()
plt.imshow(nuclei)
plt.savefig('E:/wulingfei/image_processing/dna_6.jpeg', bbox_inches=None)
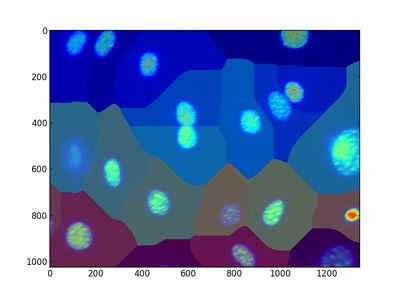
我们当然也可以把区域划分图与之前的原始图像重叠在一起:
plt.imshow(dna,alpha=0.6)
plt.savefig('E:/wulingfei/image_processing/dna_7.jpeg', bbox_inches=None)
对人物图像的处理
原始数据及预处理
先调用一些常用包
import mahotas as mh
from matplotlib import pyplot as plt
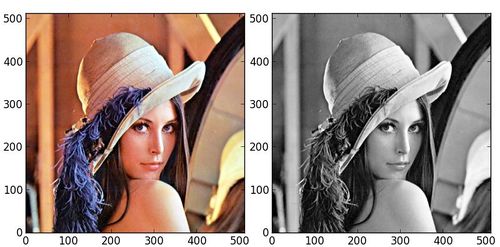
读入图后转变为灰度图
#---to gray scale--------
figure(num=None, figsize=(8, 4), dpi=80, facecolor='w', edgecolor='k')
plt.subplot(1,2,1)
image = mh.imread('E:/wulingfei/image_processing/Lenna_raw.jpg')
plt.imshow(image,origin='lower')
plt.subplot(1,2,2)
imgray = mh.colors.rgb2gray(image, dtype=np.uint8)
plt.gray()
plt.imshow(imgray,origin='lower')
plt.tight_layout(pad=0.4, w_pad=0, h_pad=1.0)
plt.savefig('E:/wulingfei/image_processing/lenna_1.jpeg', bbox_inches=None)
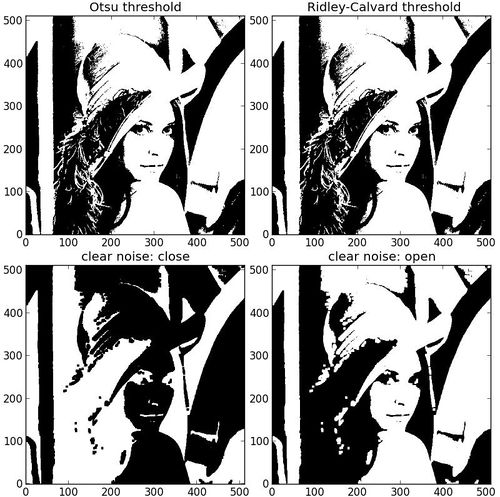
使用阈值处理的方法区分图片不同区域
选定阈值,使用阈值做判定,将像素矩阵转变为01矩阵。
figure(num=None, figsize=(8, 8), dpi=80, facecolor='w', edgecolor='k')
plt.subplot(2,2,1)
T = mh.thresholding.otsu(imgray)
bimage = (imgray > T)
plt.gray()
plt.imshow(bimage,origin='lower')
plt.title("Otsu threshold")
plt.subplot(2,2,2)
T2 = mh.thresholding.rc(imgray)
bimage = (imgray > T)
plt.gray()
plt.imshow(bimage,origin='lower')
plt.title(" Ridley-Calvard threshold")
plt.subplot(2,2,3)
otsubin = (imgray <= T)
otsubin = mh.close(otsubin, np.ones((5,5)))
plt.imshow(otsubin,origin='lower' )
plt.title("clear noise: close")
plt.subplot(2,2,4)
otsubin = (imgray > T)
otsubin = mh.open(otsubin, np.ones((5,5)))
plt.title("clear noise: open")
plt.imshow(otsubin,origin='lower' )
plt.tight_layout(pad=0.4, w_pad=0, h_pad=1.0)
plt.savefig('E:/wulingfei/image_processing/lenna_2.jpeg', bbox_inches=None)
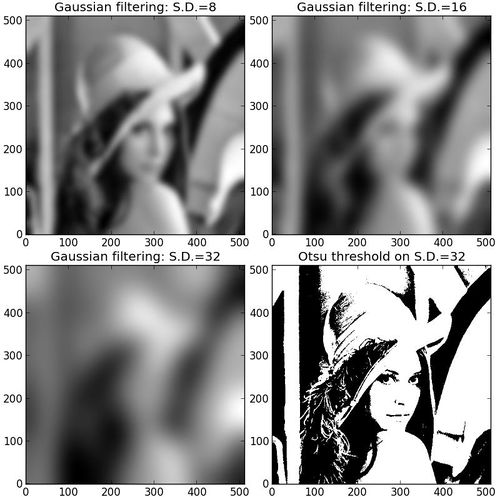
高斯过滤极其阈值效果
使用高斯矩阵与像素矩阵做卷积。
figure(num=None, figsize=(8, 8), dpi=80, facecolor='w', edgecolor='k')
plt.subplot(2,2,1)
im8 = mh.gaussian_filter(imgray,8)
plt.imshow(im8,origin='lower' )
plt.title("Gaussian filtering: S.D.=8")
plt.subplot(2,2,2)
im16 = mh.gaussian_filter(imgray,16)
plt.imshow(im16,origin='lower' )
plt.title("Gaussian filtering: S.D.=16")
plt.subplot(2,2,3)
im32 = mh.gaussian_filter(imgray,32)
plt.imshow(im32,origin='lower' )
plt.title("Gaussian filtering: S.D.=32")
plt.subplot(2,2,4)
T = mh.thresholding.otsu(im32)
bimage = (imgray > T)
plt.gray()
plt.imshow(bimage,origin='lower')
plt.title("Otsu threshold on S.D.=32")
plt.tight_layout(pad=0.4, w_pad=0, h_pad=1.0)
plt.savefig('E:/wulingfei/image_processing/lenna_3.jpeg', bbox_inches=None)
怀旧风格
制造白色和黑色的随机噪声,覆盖在灰度图上。
#------Adding salt and pepper noise-------
figure(num=None, figsize=(8, 8), dpi=80, facecolor='w', edgecolor='k')
plt.subplot(2,2,1)
plt.imshow(imgray,origin='lower')
plt.title("raw")
plt.subplot(2,2,2)
salt = np.random.random(imgray.shape) > .975
pepper = np.random.random(imgray.shape) > .975
plt.imshow(salt)
plt.title("salt/pepper distribution")
plt.subplot(2,2,3)
imsalt=np.maximum(salt*200, imgray)
plt.imshow(imsalt,origin='lower')
plt.title("adding salt")
plt.subplot(2,2,4)
impeper=np.minimum(pepper*30 + imsalt*(~pepper), imsalt)
plt.imshow(impeper,origin='lower')
plt.title("adding pepper")
plt.tight_layout(pad=0.4, w_pad=0, h_pad=1.0)
plt.savefig('E:/wulingfei/image_processing/lenna_4.jpeg', bbox_inches=None)
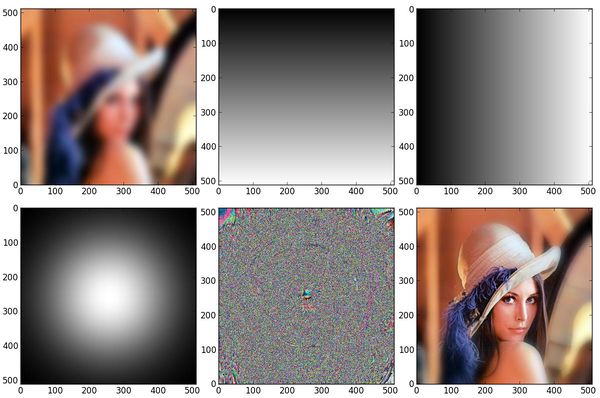
bokeh(背景虚化)技巧
先像素矩阵分割成红蓝绿三个通道,各自进行高斯过滤,再组合在一起。构建清晰度权重,矩阵中间最高,边缘最低,使用该权重对组合得到的图像进行加权。
#------Putting the center in focus-------
figure(num=None, figsize=(12, 8), dpi=80, facecolor='w', edgecolor='k')
# This breaks up the image into RGB channels
r, g, b = image.transpose(2, 0, 1)
h, w = r.shape
# smooth the image per channel:
r12 = mh.gaussian_filter(r, 12.)
g12 = mh.gaussian_filter(g, 12.)
b12 = mh.gaussian_filter(b, 12.)
# build back the RGB image
im12 = mh.as_rgb(r12, g12, b12)
plt.subplot(2,3,1)
plt.imshow(im12,origin='lower')
X, Y = np.mgrid[:h, :w]
X = X - h / 2.
Y = Y - w / 2.
X /= X.max()
Y /= Y.max()
plt.gray()
plt.subplot(2,3,2)
plt.imshow(X)
plt.subplot(2,3,3)
plt.imshow(Y)
# Array C will have the highest values in the center, fading out to the edges:
C = np.exp(-2. * (X ** 2 + Y ** 2))
plt.subplot(2,3,4)
plt.imshow(C)
C -= C.min()
C /= C.ptp()
C = C[:, :, None]
plt.subplot(2,3,5)
newdata=image * C + (1 - C) * im12
plt.imshow(newdata,origin='lower')
# The final result is sharp in the centre and smooths out to the borders:
plt.subplot(2,3,6)
ring = mh.stretch(newdata)
plt.imshow(ring,origin='lower')
plt.tight_layout(pad=0.4, w_pad=0, h_pad=1.0)
plt.savefig('E:/wulingfei/image_processing/lenna_5.jpeg', bbox_inches=None)
波普艺术
在矩阵上加一些随机噪声
figure(num=None, figsize=(12, 4), dpi=80, facecolor='w', edgecolor='k')
plt.subplot(1,3,1)
plt.axis('off')
plt.imshow(image+0.23,origin='lower')
plt.subplot(1,3,2)
plt.axis('off')
plt.imshow(image+0.53,origin='lower')
plt.subplot(1,3,3)
plt.axis('off')
plt.imshow(image+0.73,origin='lower')
plt.tight_layout(pad=0.4, w_pad=0, h_pad=1.0)
plt.savefig('E:/wulingfei/image_processing/lenna_6.jpeg', bbox_inches=None)