新陈代谢
新陈代谢(Metabolism /məˈtæbəlɪzəm/,来自希腊语:μεταβολή metabolē,“变化”)是生物体内维持生命的化学反应。新陈代谢的三个主要目的是:将食物转化为能量以运行细胞过程;将食物或燃料转化为蛋白质、脂类、核酸和一些碳水化合物的构件;以及消除代谢废物。这些酶催化的反应使生物体得以生长和繁殖,维持其结构,并对其环境作出反应。(新陈代谢这个词也可以指生物体内发生的所有化学反应的总和,包括消化和物质在细胞内以及不同细胞之间的运输,在这种情况下,上述细胞内的一系列反应称为中间代谢或中级代谢)。
新陈代谢反应可分为分解代谢-分解化合物(例如,通过细胞呼吸将葡萄糖分解为丙酮酸) ; 或合成代谢-合成化合物(例如蛋白质、碳水化合物、脂类和核酸)。通常,分解代谢释放能量,合成代谢消耗能量。
新陈代谢的化学反应被组织成代谢途径,在代谢途径中,一种化学物质通过一系列步骤转化为另一种化学物质,每一步都由特定的酶来推动。酶对新陈代谢至关重要,因为它们通过将生物体与释放能量的自发反应耦合,使生物体能够驱动需要能量的理想反应,而这些反应本身不会发生。酶起催化剂的作用——它们使反应进行得更快——它们还可以调节代谢反应的速率,例如对细胞环境的变化或其他细胞发出的信号作出反应。
特定生物体的新陈代谢系统决定了哪些物质有营养,哪些有毒。例如,一些原核生物利用硫化氢作为营养物质,然而这种气体对动物是有毒的[1]。生物体的基础代谢率是所有这些化学反应所消耗能量的量度。
新陈代谢的一个显著特征是,不同物种之间的基本新陈代谢途径具有相似性[2]。例如,作为三羧酸循环中的中间体,最著名的一组羧酸存在于所有已知的生物体中,它们在单细胞细菌大肠杆菌和巨大的多细胞生物(如大象)中都能被找到[3]。 这些代谢途径的相似性很可能是由于它们在演化史的早期出现,然后又因为它们的功效而保留下来[4][5]。癌细胞的代谢也不同于正常细胞的代谢,这些差异可以用来寻找癌细胞治疗的靶点[6]。
关键的生物化学成分
构成动物、植物和微生物的大部分结构由四种基本分子组成: 氨基酸、糖类化合物、核酸和脂类(通常称为脂肪)。由于这些分子对生命至关重要,新陈代谢反应要么专注于在构建细胞和组织的过程中制造这些分子,要么将这些分子作为能量来源并将其消化分解。这些生化物质可以结合在一起形成DNA和蛋白质之类的聚合物,它们都是生命必不可少的大分子聚合物[7]。
| 分子类型 | 单体形式的名称 | 聚合物形式的名称 | 聚合物形态的例子 |
|---|---|---|---|
| 氨基酸 | 氨基酸 | 蛋白质(由多肽组成) | 纤维蛋白和球状蛋白 |
| 碳水化合物 | 单糖 | 多糖 | 淀粉、糖原和纤维素 |
| 核酸 | 核苷酸 | 多核苷酸 | DNA和RNA |
氨基酸和蛋白质
蛋白质是由氨基酸组成的线性链,它们通过肽键连接在一起。许多蛋白质是在新陈代谢中催化化学反应的酶。其他蛋白质具有结构或机械功能,例如那些形成细胞骨架的蛋白质(细胞骨架是维持细胞形状的支架系统)[8] 。蛋白质在细胞信号传导、免疫反应、细胞粘附、主动跨膜转运和细胞周期中也很重要[9] 。氨基酸还通过提供碳源进入细胞三羧酸循环,促进细胞的能量代谢,[10]尤其是在葡萄糖等主要能量来源匮乏或细胞发生代谢应激时[11]。
脂类
脂类是最多样化的生物化学物质。它们的主要结构用途是作为生物膜内部和外部的一部分,如细胞膜,或作为能量来源[9]。脂类通常被定义为疏水性或两亲性的生物分子,但会溶解在有机溶剂中,如酒精、苯或氯仿。[12]脂肪是一大类含有脂肪酸和甘油的化合物,一个甘油分子连接到三个脂肪酸酯即称为三酰甘油酯。[13]这种基本结构存在一些变异,包括主骨(如鞘磷脂中到鞘氨醇)和亲水基(如磷脂中的磷酸盐)。类固醇,如固醇,是另一类主要的脂类[14]。
碳水化合物
碳水化合物是醛或酮,带有许多羟基,能以直链或环的形式存在。碳水化合物是最丰富的生物分子,承担着许多作用,如能量的储存和运输(淀粉、糖原)和作为结构要件(植物的纤维素、动物的甲壳素)。基本的碳水化合物单位称为单糖[9],包括半乳糖、果糖以及最重要的葡萄糖。单糖能以几乎无限多样的方式连接在一起形成多糖[15]。
核苷酸
DNA和RNA这两种核酸是核苷酸的聚合物。每个核苷酸都是由一个磷酸连接到核糖或脱氧核糖糖基上形成的,而核糖或脱氧核糖糖基又连接到含氮碱基上。核酸对于遗传信息的储存和使用,以及通过转录和蛋白质生物合成过程对其进行解释至关重要。这些信息受到DNA修复机制的保护[9],并通过DNA复制进行传播。许多病毒都有RNA基因组,如HIV病毒,它利用逆转录从其病毒RNA基因组中创建DNA模板。核糖体和核糖体等核糖体中的RNA类似于酶[16],因为它可以催化化学反应。单个核苷是通过将核碱基连接到核糖上制成的。这些碱基是含氮的杂环,分为嘌呤或嘧啶。核苷酸还在代谢基团转移反应中充当辅酶[17]。
辅酶
新陈代谢涉及大量的化学反应,但大多数属于几种基本类型的反应,涉及原子的官能团及其键在分子内的转移。这种常见的化学反应使细胞能够用一小套代谢中间体在不同反应之间携带化学基团[18]。这些基团转移中间体称为辅酶[17]。每一类基团转移反应都是由一种特定的辅酶进行的,它是一组产生它的酶和消耗它的酶的底物。因此,这些辅酶不断地被制造、消耗,然后循环利用[19]。
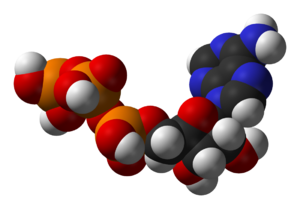
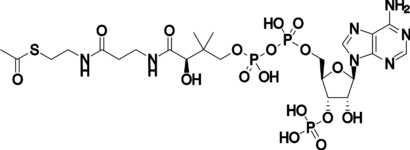
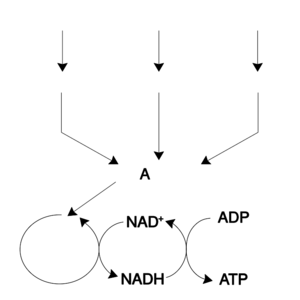
其中一种中心辅酶是三磷酸腺苷(ATP),它是细胞的通用能源货币。这种核苷酸在不同的化学反应之间传递化学能。细胞中只有少量的ATP,但由于ATP是不断再生的,所以人体每天可以使用大约相当于自身重量的ATP[19]。ATP是分解代谢和合成代谢之间的桥梁。分解代谢分解分子,合成代谢则将它们组合在一起。分解反应产生ATP,合成代谢反应则消耗ATP。它也是磷酸化反应中磷酸基团的载体[20]。
维生素是一类细胞不能合成的微量有机化合物。在人体营养中,大多数维生素经过修饰后都具有辅酶的功能,例如,所有水溶性维生素在细胞中使用时都会被磷酸化或与核苷酸偶联[21]。烟酰胺腺嘌呤二核苷酸(NAD+)是维生素B3(烟酸)的衍生物,它是一种重要的辅酶,起着氢接受器的作用。数百种不同类型的脱氢酶从其底物中去除电子,并将NAD+还原成NADH。这种还原形式的辅酶是细胞中任何需要还原其底物的还原酶的底物。烟酰胺腺嘌呤二核苷酸在细胞中以两种相关形式存在[22],即NADH和NADPH。NAD +/NADH 形式在分解代谢反应中起重要作用,而 NADP +/NADPH 形式在分解代谢反应中起重要作用[23]。
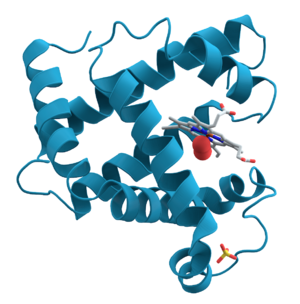
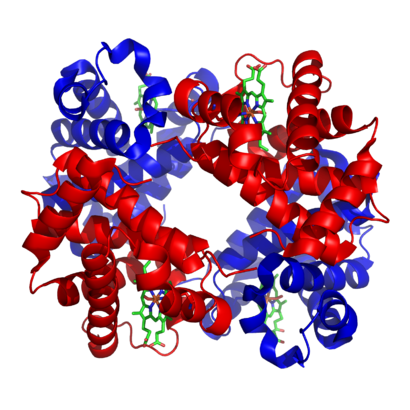
. 蛋白质亚基为红色和蓝色,含铁血红素基为绿色。]]
矿物质和辅因子
无机元素在新陈代谢中起着关键作用; 有些元素含量丰富(例如:钠和钾) ,而另一些元素则在微量浓度下发挥作用。人的体重约99%是由碳、氮、钙、钠、氯、钾、氢、磷、氧和硫等元素组成。有机化合物(蛋白质、脂类和碳水化合物)含有大部分的碳和氮;大部分的氧和氢以水的形式存在。
丰富的无机元素可以电解质。最重要的离子是钠、钾、钙、镁、氯化物、磷酸盐和有机离子重碳酸盐。维持细胞膜上精确的离子梯度可以维持渗透压和 ph 值。离子对于神经和肌肉功能也是至关重要的[24],因为这些组织中的动作电位是由细胞外液和细胞液之间的电解质交换产生的。电解质通过细胞膜上称为离子通道的蛋白质进入和离开细胞[25] 。例如,肌肉收缩依赖于钙、钠和钾通过细胞膜和T管道中的离子通道的运动[26]。
过渡金属通常以微量元素的形式存在于生物体内,其中锌和铁最为丰富[27]。这些金属元素在某些蛋白质中作为辅因子,是催化酶等酶和血红蛋白等氧载体蛋白活性所必需的金属辅因子。[28]金属辅因子与蛋白质中的特定位点紧密结合,虽然在催化过程中酶的辅因子可以被改变,但在催化反应结束时,它们总是恢复到原来的状态。金属微量营养素由特定的转运体带入生物体内,不用时与储存蛋白如铁蛋白或金属硫蛋白结合[29][30]。
分解代谢
分解代谢是指分解大分子的一系列代谢过程。其中包括分解和氧化食物分子。分解代谢反应的目的是为构建分子的合成代谢反应提供所需的能量和成分。这些分解代谢反应的确切性质因生物体而异[31],生物体可以根据它们的能量和碳的来源(其主要营养组)进行分类,如下表所示。有机养生物把有机分子作为能量来源,而岩养生物利用无机基质,光养生物利用阳光(获得化学能)[32] 。然而,所有这些不同形式的新陈代谢都依赖于氧化还原反应,这些反应涉及电子从还原的供体分子(如有机分子,水,氨,硫化氢或亚铁离子)转移到受体分子(如氧,硝酸盐或硫酸盐)。在动物中,这些反应涉及复杂的有机分子,它们被分解成更简单的分子,如二氧化碳和水。在诸如植物和蓝藻这样的光合生物体中,这些电子转移反应不释放能量,而是用来储存从阳光中吸收的能量[33]。
| 能量源 | 阳光 | photo- | -troph | ||
| 预生成分子 | chemo- | ||||
| 电子供体 | 有机化合物 | organo- | |||
| 无机化合物 | litho- | ||||
| 碳源 | 有机化合物 | hetero- | |||
| 无机化合物 | auto- | ||||
动物中最常见的分解代谢反应可以分为三个主要阶段。在第一阶段,大的有机分子,如蛋白质,多糖或脂类,在细胞外被消化成较小的分子。接下来,这些较小的分子被细胞吸收并转化成更小的分子,通常是乙酰辅酶A (acetyl-CoA) ,并释放出一些能量。最后,辅酶 A 上的乙酰基在三羧酸循环和电子传递链中被氧化成水和二氧化碳,释放出储存的能量,将辅酶烟酰胺腺嘌呤二核苷酸(NAD+)还原成NADH[31]。
消化
大分子不能直接被细胞处理。大分子必须先被分解成较小的单位,才能用于细胞代谢。不同类别的酶被用来消化这些聚合物。这些消化酶包括将蛋白质消化成氨基酸的蛋白酶,以及将多糖消化成单糖的糖苷水解酶[35]。
微生物简单直接地将消化酶分泌到周围环境中[36][37],而动物必须通过它们肠道(包括胃、胰腺和唾液腺)中的特定细胞分泌这些酶。[38]这些细胞外酶释放的氨基酸或糖通过活性转运蛋白被泵入细胞内[39][40]。
有机化合物的能量
碳水化合物分解代谢是将碳水化合物分解成较小的单位的过程。碳水化合物一旦被消化成单糖,通常就被带入细胞。一旦进入细胞内[41],分解的主要途径就是糖酵解,其中糖(例如葡萄糖和果糖)被转化为丙酮酸并生成一些ATP。[42]丙酮酸是几种代谢途径中的中间体,但大多数通过有氧(含氧)糖酵解转化为乙酰辅酶A并进入三羧酸循环。尽管在三羧酸循环中会产生更多的ATP,但最重要的产物是NADH,它是由 NAD < sup > + 在乙酰辅酶A被氧化后制成的。这种氧化释放出作为废物的二氧化碳。在厌氧条件下,糖酵解产生乳酸盐,即由乳酸脱氢酶将丙酮酸盐转化为乳酸盐,同时将NADH重新氧化为NAD < sup > + 再用于糖酵解。[43]葡萄糖分解的另一种途径是磷酸戊糖途径,它还原辅酶NADPH并产生戊糖,如核糖(核酸的糖成分)。
脂肪通过水解作用分解为游离脂肪酸和甘油。甘油进入糖酵解,脂肪酸被 β氧化分解,释放出乙酰辅酶A,然后进入三羧酸循环。脂肪酸在氧化时会释放比碳水化合物更多的能量,因为碳水化合物的结构中含有更多的氧。类固醇也会被一些细菌在类似于 β 氧化的过程中分解,这个分解过程会释放出大量的乙酰辅酶A、丙酰辅酶A和丙酮酸,它们都可以给细胞提供能量。结核杆菌也可以依靠脂质胆固醇这唯一的碳源生长,而且参与胆固醇利用途径的基因已经被证实在结核杆菌感染生命周期的不同阶段都是重要的[44]。
氨基酸可以用来合成蛋白质和其他生物分子,也可以被氧化成尿素和二氧化碳从而提供能量[45]。氧化途径从转氨酶去除氨基酸上的氨基开始。氨基进入尿素循环,留下酮酸形式的脱氨基碳骨架。其中一些酮酸是三羧酸循环的中间产物,例如谷氨酸的脱氨反应形成 α- 酮戊二酸[46]。葡萄糖原氨基酸也可以通过糖异生作用转化为葡萄糖(具体内容见下文)[47]。
能量转换
氧化磷酸化
氧化磷酸化中,通过如柠檬酸循环等代谢途径,电子从被消化吸收的食物分子上转移到氧气上,并将产生的能量以ATP的方式储存起来。在真核生物中,这一过程是通过线粒体膜上的一系列膜蛋白来完成的,被称为电子传递链。而在原核生物中,这些蛋白质存在于细胞的内膜中。[48]这些蛋白质利用电子从还原性分子(如NADH)传递到氧气所释放的能量来泵送质子穿过细胞膜[49]。
将质子泵出线粒体,会在膜上形成质子浓度差,产生电化学梯度。[50]这种力量促使质子通过ATP合成酶的基座回到线粒体中。质子的流动使柄亚基旋转,从而改变合成酶域的活性位点的形状,使二磷酸腺苷磷酸化--变成ATP[19]。
无机化合物的能量
化能无机营养是在原核生物中发现的一种新陈代谢,其能量来自于无机化合物的氧化。这些生物可以利用氢气、[51] 还原硫化合物(如硫化物、硫化氢和硫代硫酸酯[52])或氨[53]作为还原力的来源,它们从这些化合物与氧或亚硝酸盐等电子接受体的氧化作用中获得能量。这些微生物过程[54]在全球生物地球化学循环(如乙酰化、硝化和反硝化)中非常重要,对土壤肥力也很关键[55][56]。
光能
阳光中的能量被植物、蓝藻、紫细菌、绿硫细菌和一些原生生物所吸收。这个过程通常与二氧化碳转化为有机化合物相结合,这是光合作用的一部分,下文将对此进行讨论。然而,原核生物的能量捕获和碳固定系统可以单独运作,因为紫色细菌和绿硫细菌可以利用阳光作为能源,同时在碳固定和有机化合物发酵之间转换[57][58]。
在许多生物体中,太阳能的获取在原理上类似于氧化磷酸化,因为它涉及到以质子浓度梯度的形式储存能量。这种质子动力驱动 ATP 的合成[59] 。驱动这种电子传递链所需的电子来自于聚光蛋白质,这种蛋白质被叫做光合反应中心。根据存在的光合色素的性质,反应中心分为两种类型。大多数光合细菌只有一种类型,而植物和蓝藻有两种类型[60]。
在植物、藻类和蓝藻中,光系统 II 利用光能将电子从水中移走,释放出氧气。然后电子流向细胞色素b6f蛋白复合体,后者利用它们的能量穿过叶绿体中的类囊体膜,泵入质子[33]。这些质子在驱动ATP合成酶时通过膜向后移动,就像之前一样。然后电子流经光系统I,可以用来减少辅酶NADP< sup > + [61]。这些辅酶可用于卡尔文循环(下文将对此进行讨论),或被循环用于进一步生成ATP。
合成代谢
合成代谢是一系列建设性的新陈代谢过程,在这一系列过程中分解代谢所释放的能量被用来合成复杂的分子。一般来说,组成细胞结构的复杂分子是由小而简单的前体逐步构成的。合成代谢包括三个基本阶段。首先是氨基酸、单糖、异戊二烯和核苷酸等前体的产生,其次是利用ATP产生的能量将它们活化成活性形式,第三是将这些前体组装成复杂的分子,如蛋白质、多糖、脂质和核酸[62]。
生物体的合成代谢根据其细胞中构建分子的来源而有所不同。植物等自养生物可以在细胞中利用二氧化碳和水等简单分子中构建复杂的有机分子(如多糖和蛋白质)。而异养生物则需要更复杂的物质来源(如单糖和氨基酸)才能产生这些复杂的分子。生物可以根据其能量的最终来源进一步分类: 光自养生物和光异养生物从光中获得能量,而化能自养生物和化能异养生物从无机氧化反应中获得能量[62]。
碳固定
光合作用就是依靠阳光和二氧化碳(CO2)合成碳水化合物。在植物中,蓝细菌和藻类的含氧光合作用使水分解,排出氧气。如前所述,这一过程利用光合反应中心产生的ATP和NADPH将CO2转化为三磷酸甘油酯,然后再转化为葡萄糖。这个固碳反应作为卡尔文-本森Calvin – Benson 循环的一部分是在RuBisCO酶的催化下进行的[63]。植物有三种类型的光合作用:C3固碳、C4固碳和CAM光合作用。它们的不同之处在于二氧化碳进入卡尔文循环的路径不同,C3植物直接固定二氧化碳,而C4和CAM植物首先将二氧化碳吸收到其他化合物中,以适应强烈的阳光和干燥的环境[64]。
在能够光合作用的原核生物中,碳固定的机制更加多样。对它们而言,二氧化碳可以通过Calvin– Benson循环、反向三羧酸循环[65]或乙酰辅酶A的羧化作用得到固定。原核化能自养生物也通过Calvin– Benson环固定CO2 [66][67],但它们利用无机化合物的能量来驱动反应[68]。
碳水化合物和聚糖
在碳水化合物合成代谢过程中,简单的有机酸可转化为葡萄糖等单糖,再用于合成淀粉等多糖。由丙酮酸、乳酸、甘油、3-磷酸甘油酸和氨基酸等化合物生成葡萄糖称为葡萄糖异生。糖异生作用通过一系列中间产物将丙酮酸转化为葡萄糖-6-磷酸,其中许多中间产物与糖酵解过程相同[42]。然而,这一途径并不是简单的糖酵解逆向运行,因为有几个步骤是由非糖酵解酶催化的。这很重要,因为它使得葡萄糖的形成和分解可以分别被调节,而且防止了两条途径在无效循环中同时运行[69][70]。
虽然脂肪是储存能量的一种常见方式,但在脊椎动物(如人类)体内储存的脂肪酸不能通过葡萄糖异生作用转化为葡萄糖,因为这些生物不能将乙酰辅酶A转化为丙酮酸;植物有必要的酶催化机制,而动物没有[71]。因此,在长期饥饿后,脊椎动物需要从脂肪酸中产生酮体,以取代大脑等不能代谢脂肪酸的组织中的葡萄糖。在其他生物体如植物和细菌中,这个代谢问题是靠乙醛酸循环来解决的[72] 。乙醛酸循环绕过三羧酸循环中的脱羧步骤,并将乙酰辅酶a转化为草酰乙酸,在那里它可以用来生产葡萄糖。除了脂肪[71][73],葡萄糖也作为一种能量资源储存在大多数组织中,一般会通过它的糖化来维持血液中的葡萄糖水平[74] 。
多糖和聚糖是在糖基转移酶作用下,将单糖从活性糖-磷酸盐供体(如尿苷二磷酸葡萄糖(UDP-Glc))依次加入到生长中的多糖的受体羟基上形成的。由于底物环上的任何羟基都可以作为受体,所以产生的多糖会有直链或支链结构[75]。产生的多糖本身具有结构或代谢功能,还可以通过低聚糖转移酶转移到脂质和蛋白质中[76][77]。
脂肪酸,类异戊二烯和固醇
脂肪酸是由脂肪酸合成酶聚合并还原乙酰辅酶A单元制成的。脂肪酸中的酰基链通过一些反应的循环得以延伸,这些反应包括:添加酰基,将其还原为醇,脱水成烯烃基团,然后再还原为烷烃基团。脂肪酸生物合成的酶可以分为两类:在动物和真菌中,所有脂肪酸合成酶的反应都是由单一的多功能I型蛋白来完成的[78],而在植物的质体和细菌中,则由单独的II型酶来完成每一步[79][80]。
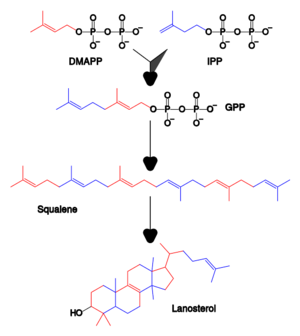
萜烯和异戊二烯是一大类脂类,包括类胡萝卜素,也是最大的一类植物天然产品[81]。这些化合物是由反应性前体焦磷酸异戊烯酯和焦磷酸二甲基烯丙基酯所提供的异戊二烯单元组装和改性而成。这些前体可以靠不同的途径制造[82]。在动物和古生物中,甲戊二酸途径从乙酰辅酶A产生这些化合物[83],而在植物和细菌中,非甲戊二酸途径使用丙酮酸和甘油醛3-磷酸作为底物。使用这些活化的异戊二烯供体的一个重要反应是固醇的生物合成[82][84]。在该反应中,异戊二烯单元连接在一起,制成角鲨烯,然后折叠起来形成一组环,制成羊毛固醇[85]。羊毛固醇随后可转化为其他固醇,如胆固醇和麦角固醇[85][86]。
蛋白质类
生物体合成这20种常见氨基酸的能力各不相同。大多数细菌和植物都能合成这20种氨基酸,但哺乳动物只能合成11种非必需氨基酸,因此必须从食物中获得9种必需氨基酸[9]。所有的氨基酸都是由糖酵解、三羧酸循环或磷酸戊糖途径的中间产物合成的[87]。氮由谷氨酸和谷氨酰胺提供。非关键氨基酸的合成取决于适当的α-酮酸的形成,它可以转氨基形成氨基酸[88]。
氨基酸通过肽键链连接成蛋白质。每种不同的蛋白质都有一个独特的氨基酸残基序列:这就是它的主要结构。就像字母表的字母可以组合成无穷无尽的各种单词一样,氨基酸也能以不同的序列连接起来,形成种类繁多的蛋白质。蛋白质是由氨基酸制成的,这些氨基酸通过酯键附着在转运RNA(tRNA)分子上而被激活。氨基酰tRNA前体是在靠氨基酰tRNA合成酶进行的ATP依赖性反应中产生的.[89]。然后,这种氨基酰tRNA成为核糖体的底物,核糖体利用信使RNA中的序列信息将氨基酸连接到伸长的蛋白质链上[90]。
核苷酸的合成和补救途径
核苷酸由氨基酸、二氧化碳和甲酸在需要大量代谢能量的途径中生成[91]。因此,大多数生物体都有有效的系统来挽救预先形成的核苷酸[91][92]。嘌呤以核苷的形式合成(碱基附着在核糖上)。腺嘌呤和鸟嘌呤都是由一磷酸核苷肌苷前体合成的[93],而前体是由甘氨酸、谷氨酰胺和天冬氨酸的原子合成的,从辅酶四氢叶酸转移来的甲酸酯也是如此。另一方面,嘧啶是由磷酸基合成的,而磷酸基是由谷氨酰胺和天门冬氨酸形成的[94]。
异种生物学和氧化还原代谢
所有的生物都不断地接触到它们不能进食的化合物,如果它们在细胞中积累这些化合物就会被伤害,因为它们没有新陈代谢功能。这些具有潜在破坏性的化合物被称为异生物质[95]。合成药物、天然毒物和抗生素等异生物质是通过一系列异生物质代谢酶来解毒的。在人体中,这些酶包括细胞色素 P450氧化酶[96]、UDP-葡萄糖醛酸转移酶[97]和谷胱甘肽 s- 转移酶。这套酶系统的作用分为三个阶段[98],首先氧化异生物质(第一阶段) ,然后将水溶性基团共轭到分子上(第二阶段)。经过降解的水溶性异生物质随后会从细胞中泵出,对多细胞生物来说,还会在排出之前进一步代谢(第三阶段)。在生态学中,这些反应在微生物对污染物的生物降解以及污染土地和溢油的生物修复中尤为重要[99]。这些微生物反应中有许多是与多细胞生物相同的,但是由于微生物种类的惊人多样性,这些微生物能够处理比多细胞生物更广泛的异生物质,甚至能够降解有机氯化合物等持久性有机污染物[100]。
对于好氧生物来说,一个相关的问题是氧化应激[101]。这个过程包括氧化磷酸化和蛋白质折叠时二硫键的形成,它产生了活性氧类(如过氧化氢)[102]。这些破坏性的氧化剂被抗氧化代谢物(如谷胱甘肽和酶)和酶(如过氧化氢酶和过氧化物酶)去除[103][104]。
生命有机体的热力学
生命有机体一定会遵守热力学定律,该定律描述了热量和功的传递。热力学第二定律指出,在任何封闭系统中,熵的总量(混乱度)不会减少。尽管生物体惊人的复杂性似乎与这一定律相矛盾,但生命是可能的,因为它们是与环境交换物质和能量的开放系统。也就是说,生命系统并不处于平衡状态,而是耗散系统,它通过大量增加环境熵来维持其高度复杂的状态[105]。细胞的新陈代谢通过将分解代谢的自发过程和合成代谢的非自发过程进行耦合来实现这一点。从热力学的角度来看,新陈代谢通过制造混乱来维持秩序[106]。
调控
由于大多数生物体的环境是不断变化的,因此它们必须对新陈代谢的反应进行精细的调节,以维持细胞内一系列恒定的条件,这种条件称为稳态[107][108]。代谢调节也使生物体能够对信号作出反应,并与环境积极互动。有两个密切相关的概念对于理解“代谢途径是如何被控制的”十分重要[109]。首先,途径中酶的调节是指其活性如何响应信号从而增加和减少。其次,这种酶所施加的控制是指它的活性变化对通路的总体速率(通过通路的通量)的影响[110]。例如,一种酶可能表现出很大的活性变化(即它是高度受控的),但如果这些变化对某一代谢途径的通量影响不大,那么这种酶就不参与该途径的控制[111]。
新陈代谢调节有多个层次。在内在调节中,代谢途径自我调节,以应对底物或产物数量的变化;例如,产物数量的减少可以增加通过该途径的通量以进行补偿[110]。外在控制是指多细胞生物体中的一个细胞根据来自其他细胞的信号改变其代谢[112]。这些信号通常以水溶性信使的形式出现(如激素和生长因子)[113],并被细胞表面的特定受体检测到。然后,这些信号通过第二信使系统在细胞内传递,该系统通常涉及蛋白质的磷酸化[114]。
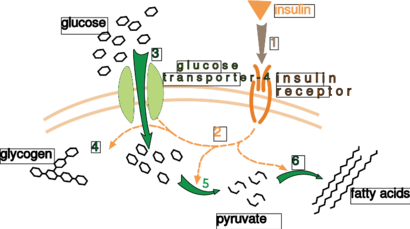
一个非常好理解的外在控制的例子是激素胰岛素对葡萄糖代谢的调节[115]。胰岛素是在血糖升高时产生的。胰岛素与细胞上的胰岛素受体结合,然后激活一连串的蛋白激酶,使细胞吸收葡萄糖,并将其转化为储存分子(如脂肪酸和糖原)[116]。糖原的新陈代谢受到分解糖原的磷酸化酶和制造糖原的糖原合成酶的活性控制。这些酶的调节方式是相互的,磷酸化作用抑制糖原合成酶,但激活磷酸化酶。胰岛素通过激活蛋白磷酸酶使这些酶的磷酸化程度降低,从而触发糖原合成[117]。
演化
上述代谢的主要途径,如糖酵解和柠檬酸循环,存在于生物的全部三个领域中,并存在于最后的普遍共同祖先中。这种普遍的祖先细胞是原核生物[3][118],也许是一种具有广泛的氨基酸、核苷酸、碳水化合物和脂质代谢的产烷菌[119][120]。在后来的进化过程中,这些古老的途径之所以得以保留,可能是因为这些反应为它们特定的代谢问题提供了最佳解决方案,这些途径(如糖酵解和三羧酸循环)以最少的步骤高效地产生它们的最终产物。最初的基于酶的代谢途径可能是嘌呤核苷酸代谢的一部分[4][5],而之前的代谢途径是远古RNA界的一部分[121]。
人们提出了许多模型来描述新代谢途径的演变机制。其中包括将新的酶顺序添加到一个短的祖传途径,整个途径的复制和分化,吸纳原有的酶或者把这些机制组装成新的反应途径。这些机制的相对重要性尚不清楚[122],但基因组研究表明,一条途径中的酶很可能有共同的祖先,这意味着许多途径是逐步进化而来的,途径中预先存在的步骤创造出了新的功能。另一种模型来自于追踪代谢网络中蛋白质结构演化的研究[123],它指出酶被普遍利用,生物体借用酶在不同的代谢途径中执行相似的功能(在 MANET 数据库中显而易见)[124]。这些征用过程导致了进化酶的嵌合[125]。第三种可能性是,新陈代谢的某些部分可能作为“模块”存在,可以在不同的途径中重复使用,并对不同的分子执行类似的功能[126]。
除了新的代谢途径的进化,演化也会导致代谢功能的丧失。例如,在某些寄生物中,一些并非生存必需的代谢过程丢失了,而预先形成的氨基酸、核苷酸和碳水化合物可能会从寄主那里被清除[127]。在内共生生物体中也可以看到类似的代谢能力下降[128]。
调查和操纵
传统上,新陈代谢的研究采用还原法,侧重于单一的新陈代谢途径。特别有价值的是在整个有机体、组织和细胞水平上使用放射性示踪剂,通过追踪携带放射性标记的中间物和产品来获知从前体到最终产品的路径[129]。然后可以纯化催化这些化学反应的酶,并研究其动力学和对抑制剂的反应。一个并行的方法是识别细胞或组织中的小分子;这些分子的完整集合被称为代谢组。总的来说,这些研究可以很好地了解简单代谢途径的结构和功能,但当应用于更复杂的系统(如一个完整细胞的代谢时),这些研究是不够的[130]。
细胞中包含数千种不同酶的代谢网络,这种复杂性概念可以在右图中看到,右图显示了43种蛋白质和40种代谢物之间的相互作用: 基因组序列提供了包含26.500个基因的列表[131]。然而,现在能够利用这些基因组数据来重建完整的生化反应网络,并产生更全面的数学模型来解释和预测它们的行为。这些模型在用于整合通过经典方法获得的途径和代谢物数据以及蛋白质组学和DNA微阵列研究的基因表达数据时特别强大[132]。利用这些技术[133],目前已经产生了一个人类代谢的模型,这将指导未来的药物发现和生化研究。这些模型现在被用于网络分析[134],将人类疾病划分为具有共同蛋白质或代谢物的群体[135][136]。
细菌代谢网络是蝴蝶结结构[137][138][139]的一个突出例子,这种架构能够输入多种营养物质,并利用相对较少的中间通货生产出大量产品和复杂的大分子。
这些信息的一个主要技术应用是代谢工程学。在应用中,酵母、植物或细菌等生物体都可经过基因改造,使它们在生物技术方面更有用处,同时有助于生产抗生素等药物或工业化学品(如1,3- 丙二醇和莽草酸)[140]。这些基因改造通常旨在减少生产产品所使用的能源,提高产量和减少废物的产生[141]。
历史
新陈代谢这一术语源于法语“ métabolisme”或古希腊语 μταβoλλ -“Metabole”意为某种改变,它源自意为“去改变”的“ Metaballein”[142]
希腊哲学
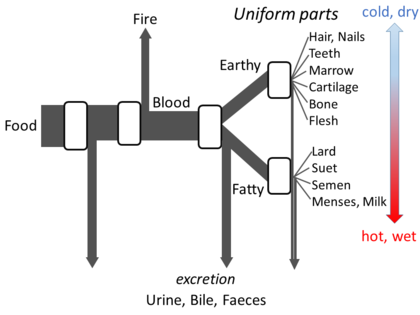
亚里士多德的《动物的各部分》详细阐述了他关于新陈代谢的观点,以便建立一个开放性的流动模型。他认为,在这个过程的每个阶段,来自食物的物质都会发生转化,释放出的热量成为火的典型元素,残余物质以尿液、胆汁或粪便的形式排出体外[143]。
伊斯兰医学
伊本·纳菲斯 Ibn al-Nafis 在其公元1260年的著作《 Al-Risalah al-Kamiliyyah fil Siera al-Nabawiyyah 》(《卡米尔关于先知传记的论述》)中描述了新陈代谢,其中包括以下短语: ”身体及其部分处于持续的溶解和营养状态,因此它们不可避免地要经历永久性的变化[144]。
科学方法的应用
新陈代谢的科学研究历史跨越了几个世纪,从早期研究中对动物整体的研究,到现代生物化学中对单个代谢反应的研究。 人类新陈代谢的第一个对照实验是由圣托里奥·桑托里奥 Santorio Santorio于1614年在他的著作《静态医学》中发表的[145]。他描述了自己在进食、睡觉、工作、性交、禁食、饮水和排泄前后的体重。他发现他摄入的大部分食物都是通过他所谓的“无知觉的汗液”流失的。
在早期的研究中,这些新陈代谢过程的机制还没有确定,人们认为一种生命力量是生命组织的活力[146]。19世纪,路易-巴斯德Louis Pasteur 在研究酵母菌将糖发酵成酒精时,得出结论:发酵是由酵母细胞内的物质催化的,他称之为 "发酵物"。他写道:"酒精发酵是与酵母细胞的生命和组织有关的行为,而与细胞的死亡或腐烂无关"。这一发现[147]连同1828年弗里德里希-沃勒Friedrich Wöhler 发表的一篇关于尿素化学合成的论文[148],因为是第一个完全由无机前体制备的有机化合物而引人注目。这证明了在细胞中发现的有机化合物和化学反应与化学的任何其他部分在原理上没有什么不同。
爱德华·布赫纳(Eduard Buchner)在20世纪初发现了酶,从而将新陈代谢的化学反应研究与细胞的生物学研究分离开来,这标志着生物化学的开始[149]。整个20世纪早期,生物化学知识的总量迅速增长。汉斯·克雷布斯(Hans Krebs)是这些现代生物化学家中最多产的一位,他对新陈代谢的研究做出了巨大的贡献。他发现了尿素循环[150],后来又与 汉斯·科恩伯格(Hans Kornberg)合作,发现了三羧酸循环和乙醛酸循环。色谱、x射线衍射、核磁共振光谱、放射性同位素标记、电子显微镜和分子动力学模拟等新技术的发展极大地助力了现代生化研究[151][73]。这些技术已经能够发现和详细分析细胞中的许多分子和代谢途径。
另请参见
- 人类新陈代谢
- 抗代谢物
- 基础代谢率
- 量热学
- 等温微量热法
- 先天性代谢缺陷
- 铁硫世界假说(生命起源的假想情景,一个"新陈代谢第一 "的生命起源理论)
- 代谢紊乱
- 微观生理学
- 主要营养组织
- 呼吸测量法
- 流代谢
- 硫代谢
- 食物的热效应
- 城市代谢
- 水代谢平衡
- 溢出代谢
- 生物途径数据库
- 京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes)
参考资料
- ↑ Friedrich C (1998). Physiology and genetics of sulfur-oxidizing bacteria. Advances in Microbial Physiology. 39. pp. 235–89. ISBN 978-0-12-027739-1.
- ↑ Pace NR (January 2001). "The universal nature of biochemistry". Proceedings of the National Academy of Sciences of the United States of America. 98 (3): 805–8. Bibcode:2001PNAS...98..805P. PMC 33372.
- ↑ 3.0 3.1 Smith E, Morowitz HJ (September 2004). "Universality in intermediary metabolism". Proceedings of the National Academy of Sciences of the United States of America. 101 (36): 13168–73. Bibcode:2004PNAS..10113168S. doi:10.1073/pnas.0404922101. PMC 516543. PMID 15340153.
- ↑ 4.0 4.1 Ebenhöh O, Heinrich R (January 2001). "Evolutionary optimization of metabolic pathways. Theoretical reconstruction of the stoichiometry of ATP and NADH producing systems". Bulletin of Mathematical Biology. 63 (1): 21–55. doi:10.1006/bulm.2000.0197. PMID 11146883.
- ↑ 5.0 5.1 Meléndez-Hevia E, Waddell TG, Cascante M (September 1996). "The puzzle of the Krebs citric acid cycle: assembling the pieces of chemically feasible reactions, and opportunism in the design of metabolic pathways during evolution". Journal of Molecular Evolution. 43 (3): 293–303. Bibcode:1996JMolE..43..293M. doi:10.1007/BF02338838. PMID 8703096.
- ↑ Vander Heiden MG, DeBerardinis RJ (February 2017). "Understanding the Intersections between Metabolism and Cancer Biology". Cell. 168 (4): 657–669. doi:10.1016/j.cell.2016.12.039. PMC 5329766. PMID 28187287.
- ↑ Cooper, Geoffrey M. (2000). "The Molecular Composition of Cells". The Cell: A Molecular Approach. 2nd Edition (in English).
- ↑ Michie KA, Löwe J (2006). "Dynamic filaments of the bacterial cytoskeleton". Annual Review of Biochemistry. 75: 467–92. doi:10.1146/annurev.biochem.75.103004.142452. PMID 16756499.
- ↑ 9.0 9.1 9.2 9.3 9.4 Nelson, David L.; Cox, Michael M. (2005). Lehninger Principles of Biochemistry. New York: W. H. Freeman and company. p. 841. ISBN 978-0-7167-4339-2. https://archive.org/details/lehningerprincip00lehn_0/page/841.
- ↑ Kelleher JK, Bryan BM, Mallet RT, Holleran AL, Murphy AN, Fiskum G (September 1987). "Analysis of tricarboxylic acid-cycle metabolism of hepatoma cells by comparison of 14CO2 ratios". The Biochemical Journal. 246 (3): 633–9. doi:10.1042/bj2460633. PMC 1148327. PMID 3120698.
- ↑ Hothersall JS, Ahmed A (2013). "Metabolic fate of the increased yeast amino Acid uptake subsequent to catabolite derepression". Journal of Amino Acids. 2013: 461901. doi:10.1155/2013/461901. PMC 3575661. PMID 23431419.
- ↑ Fahy E, Subramaniam S, Brown HA, Glass CK, Merrill AH, Murphy RC, et al. (May 2005). "A comprehensive classification system for lipids". Journal of Lipid Research. 46 (5): 839–61. doi:10.1194/jlr.E400004-JLR200. PMID 15722563.
- ↑ "Lipid nomenclature Lip-1 & Lip-2". www.qmul.ac.uk. Retrieved 2020-06-06.
- ↑ Biochemistry (8 ed.). New York: W. H. Freeman. 8 April 2015. pp. 362. ISBN 978-1-4641-2610-9.
- ↑ Raman R, Raguram S, Venkataraman G, Paulson JC, Sasisekharan R (November 2005). "Glycomics: an integrated systems approach to structure-function relationships of glycans". Nature Methods. 2 (11): 817–24. doi:10.1038/nmeth807. PMID 16278650.
- ↑ Sierra S, Kupfer B, Kaiser R (December 2005). "Basics of the virology of HIV-1 and its replication". Journal of Clinical Virology. 34 (4): 233–44. doi:10.1016/j.jcv.2005.09.004. PMID 16198625.
- ↑ 17.0 17.1 Wimmer MJ, Rose IA (1978). "Mechanisms of enzyme-catalyzed group transfer reactions". Annual Review of Biochemistry. 47: 1031–78. doi:10.1146/annurev.bi.47.070178.005123. PMID 354490.
- ↑ Mitchell P (March 1979). "The Ninth Sir Hans Krebs Lecture. Compartmentation and communication in living systems. Ligand conduction: a general catalytic principle in chemical, osmotic and chemiosmotic reaction systems". European Journal of Biochemistry. 95 (1): 1–20. doi:10.1111/j.1432-1033.1979.tb12934.x. PMID 378655.
- ↑ 19.0 19.1 19.2 Dimroth P, von Ballmoos C, Meier T (March 2006). "Catalytic and mechanical cycles in F-ATP synthases. Fourth in the Cycles Review Series". EMBO Reports. 7 (3): 276–82. doi:10.1038/sj.embor.7400646. PMC 1456893. PMID 16607397.
- ↑ Bonora M, Patergnani S, Rimessi A, De Marchi E, Suski JM, Bononi A, et al. (September 2012). "ATP synthesis and storage". Purinergic Signalling. 8 (3): 343–57. doi:10.1007/s11302-012-9305-8. PMC 3360099. PMID 22528680.
- ↑ Berg, Jeremy M.; Tymoczko, John L.; Stryer, Lubert (2002). "Vitamins Are Often Precursors to Coenzymes". Biochemistry. 5th Edition (in English).
- ↑ Pollak N, Dölle C, Ziegler M (March 2007). "The power to reduce: pyridine nucleotides--small molecules with a multitude of functions". The Biochemical Journal. 402 (2): 205–18. doi:10.1042/BJ20061638. PMC 1798440. PMID 17295611.
- ↑ Fatih, Yildiz (2009). Advances in food biochemistry. Boca Raton: CRC Press. pp. 228. ISBN 978-1-4200-0769-5.
- ↑ "Electrolyte Balance". Anatomy and Physiology. OpenStax. https://opentextbc.ca/anatomyandphysiology/chapter/26-3-electrolyte-balance/.
- ↑ Lodish, Harvey; Berk, Arnold; Zipursky, S. Lawrence; Matsudaira, Paul; Baltimore, David; Darnell, James (2000). "The Action Potential and Conduction of Electric Impulses" (in en). Molecular Cell Biology (4th ed.). https://www.ncbi.nlm.nih.gov/books/NBK21668/.
- ↑ Dulhunty AF (September 2006). "Excitation-contraction coupling from the 1950s into the new millennium". Clinical and Experimental Pharmacology & Physiology. 33 (9): 763–72. doi:10.1111/j.1440-1681.2006.04441.x. PMID 16922804.
- ↑ "Zinc Efflux in Trichomonas vaginalis: In Silico Identification and Expression Analysis of CDF-Like Genes" (in en). Quantitative Models for Microscopic to Macroscopic Biological Macromolecules and Tissues. Cham: Springer International Publishing. 2018. pp. 149–168. ISBN 978-3-319-73975-5.
- ↑ Craig Will, Leonard Ashley (2019). Manufacturing Engineering & Technology. Waltham Abbey: Scientific e-Resources. pp. 190–196. ISBN 9781839472428.
- ↑ Cousins RJ, Liuzzi JP, Lichten LA (August 2006). "Mammalian zinc transport, trafficking, and signals". The Journal of Biological Chemistry. 281 (34): 24085–9. doi:10.1074/jbc.R600011200. PMID 16793761.
- ↑ Dunn LL, Suryo Rahmanto Y, Richardson DR (February 2007). "Iron uptake and metabolism in the new millennium". Trends in Cell Biology. 17 (2): 93–100. doi:10.1016/j.tcb.2006.12.003. PMID 17194590.
- ↑ 31.0 31.1 Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (2002). "How Cells Obtain Energy from Food" (in en). Molecular Biology of the Cell (4th ed.). https://www.ncbi.nlm.nih.gov/books/NBK26882/.
- ↑ Raven, Ja (2009-09-03). "Contributions of anoxygenic and oxygenic phototrophy and chemolithotrophy to carbon and oxygen fluxes in aquatic environments". Aquatic Microbial Ecology (in English). 56: 177–192. doi:10.3354/ame01315. ISSN 0948-3055.
- ↑ 33.0 33.1 Nelson N, Ben-Shem A (December 2004). "The complex architecture of oxygenic photosynthesis". Nature Reviews. Molecular Cell Biology. 5 (12): 971–82. doi:10.1038/nrm1525. PMID 15573135.
- ↑ Brock Mikrobiologie (11., überarb. Aufl ed.). München: Pearson Studium. 2006. pp. 604, 621. ISBN 3-8273-7187-2.
- ↑ Demirel, Yaşar (2016). Energy : production, conversion, storage, conservation, and coupling (Second ed.). Lincoln: Springer. pp. 431. ISBN 978-3-319-29650-0.
- ↑ Häse CC, Finkelstein RA (December 1993). "Bacterial extracellular zinc-containing metalloproteases". Microbiological Reviews. 57 (4): 823–37. doi:10.1128/MMBR.57.4.823-837.1993. PMC 372940. PMID 8302217.
- ↑ Gupta R, Gupta N, Rathi P (June 2004). "Bacterial lipases: an overview of production, purification and biochemical properties". Applied Microbiology and Biotechnology. 64 (6): 763–81. doi:10.1007/s00253-004-1568-8. PMID 14966663.
- ↑ Hoyle T (1997). "The digestive system: linking theory and practice". British Journal of Nursing. 6 (22): 1285–91. doi:10.12968/bjon.1997.6.22.1285. PMID 9470654.
- ↑ Souba WW, Pacitti AJ (1992). "How amino acids get into cells: mechanisms, models, menus, and mediators". JPEN. Journal of Parenteral and Enteral Nutrition. 16 (6): 569–78. doi:10.1177/0148607192016006569. PMID 1494216.
- ↑ Barrett MP, Walmsley AR, Gould GW (August 1999). "Structure and function of facilitative sugar transporters". Current Opinion in Cell Biology. 11 (4): 496–502. doi:10.1016/S0955-0674(99)80072-6. PMID 10449337.
- ↑ Bell GI, Burant CF, Takeda J, Gould GW (September 1993). "Structure and function of mammalian facilitative sugar transporters". The Journal of Biological Chemistry. 268 (26): 19161–4. PMID 8366068.
- ↑ 42.0 42.1 Bouché C, Serdy S, Kahn CR, Goldfine AB (October 2004). "The cellular fate of glucose and its relevance in type 2 diabetes". Endocrine Reviews. 25 (5): 807–30. doi:10.1210/er.2003-0026. PMID 15466941.
- ↑ Alfarouk KO, Verduzco D, Rauch C, Muddathir AK, Adil HH, Elhassan GO, et al. (18 December 2014). "Glycolysis, tumor metabolism, cancer growth and dissemination. A new pH-based etiopathogenic perspective and therapeutic approach to an old cancer question". Oncoscience. 1 (12): 777–802. doi:10.18632/oncoscience.109. PMC 4303887. PMID 25621294.
- ↑ Wipperman MF, Sampson NS, Thomas ST (2014). "Pathogen roid rage: cholesterol utilization by Mycobacterium tuberculosis". Critical Reviews in Biochemistry and Molecular Biology. 49 (4): 269–93. doi:10.3109/10409238.2014.895700. PMC 4255906. PMID 24611808.
- ↑ Sakami W, Harrington H (1963). "Amino Acid Metabolism". Annual Review of Biochemistry. 32: 355–98. doi:10.1146/annurev.bi.32.070163.002035. PMID 14144484.
- ↑ Brosnan JT (April 2000). "Glutamate, at the interface between amino acid and carbohydrate metabolism". The Journal of Nutrition. 130 (4S Suppl): 988S–90S. doi:10.1093/jn/130.4.988S. PMID 10736367.
- ↑ Young VR, Ajami AM (September 2001). "Glutamine: the emperor or his clothes?". The Journal of Nutrition. 131 (9 Suppl): 2449S–59S, discussion 2486S–7S. doi:10.1093/jn/131.9.2449S. PMID 11533293.
- ↑ Hosler JP, Ferguson-Miller S, Mills DA (2006). "Energy transduction: proton transfer through the respiratory complexes". Annual Review of Biochemistry. 75: 165–87. doi:10.1146/annurev.biochem.75.062003.101730. PMC 2659341. PMID 16756489.
- ↑ Schultz BE, Chan SI (2001). "Structures and proton-pumping strategies of mitochondrial respiratory enzymes" (PDF). Annual Review of Biophysics and Biomolecular Structure. 30: 23–65. doi:10.1146/annurev.biophys.30.1.23. PMID 11340051.
- ↑ Capaldi RA, Aggeler R (March 2002). "Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor". Trends in Biochemical Sciences. 27 (3): 154–60. doi:10.1016/S0968-0004(01)02051-5. PMID 11893513.
- ↑ Friedrich B, Schwartz E (1993). "Molecular biology of hydrogen utilization in aerobic chemolithotrophs". Annual Review of Microbiology. 47: 351–83. doi:10.1146/annurev.mi.47.100193.002031. PMID 8257102.
- ↑ Weber KA, Achenbach LA, Coates JD (October 2006). "Microorganisms pumping iron: anaerobic microbial iron oxidation and reduction". Nature Reviews. Microbiology. 4 (10): 752–64. doi:10.1038/nrmicro1490. PMID 16980937.
- ↑ Jetten MS, Strous M, van de Pas-Schoonen KT, Schalk J, van Dongen UG, van de Graaf AA, et al. (December 1998). "The anaerobic oxidation of ammonium". FEMS Microbiology Reviews. 22 (5): 421–37. doi:10.1111/j.1574-6976.1998.tb00379.x. PMID 9990725.
- ↑ Simon J (August 2002). "Enzymology and bioenergetics of respiratory nitrite ammonification". FEMS Microbiology Reviews. 26 (3): 285–309. doi:10.1111/j.1574-6976.2002.tb00616.x. PMID 12165429.
- ↑ Conrad R (December 1996). "Soil microorganisms as controllers of atmospheric trace gases (H2, CO, CH4, OCS, N2O, and NO)". Microbiological Reviews. 60 (4): 609–40. doi:10.1128/MMBR.60.4.609-640.1996. PMC 239458. PMID 8987358.
- ↑ Barea JM, Pozo MJ, Azcón R, Azcón-Aguilar C (July 2005). "Microbial co-operation in the rhizosphere". Journal of Experimental Botany. 56 (417): 1761–78. doi:10.1093/jxb/eri197. PMID 15911555.
- ↑ van der Meer MT, Schouten S, Bateson MM, Nübel U, Wieland A, Kühl M, et al. (July 2005). "Diel variations in carbon metabolism by green nonsulfur-like bacteria in alkaline siliceous hot spring microbial mats from Yellowstone National Park". Applied and Environmental Microbiology. 71 (7): 3978–86. doi:10.1128/AEM.71.7.3978-3986.2005. PMC 1168979. PMID 16000812.
- ↑ Tichi MA, Tabita FR (November 2001). "Interactive control of Rhodobacter capsulatus redox-balancing systems during phototrophic metabolism". Journal of Bacteriology. 183 (21): 6344–54. doi:10.1128/JB.183.21.6344-6354.2001. PMC 100130. PMID 11591679.
- ↑ Alberts, Bruce; Johnson, Alexander; Lewis, Julian; Raff, Martin; Roberts, Keith; Walter, Peter (2002). "Energy Conversion: Mitochondria and Chloroplasts" (in en). Molecular Biology of the Cell. 4th edition. https://www.ncbi.nlm.nih.gov/books/NBK21063/.
- ↑ Allen JP, Williams JC (October 1998). "Photosynthetic reaction centers". FEBS Letters. 438 (1–2): 5–9. doi:10.1016/S0014-5793(98)01245-9. PMID 9821949.
- ↑ Munekage Y, Hashimoto M, Miyake C, Tomizawa K, Endo T, Tasaka M, Shikanai T (June 2004). "Cyclic electron flow around photosystem I is essential for photosynthesis". Nature. 429 (6991): 579–82. Bibcode:2004Natur.429..579M. doi:10.1038/nature02598. PMID 15175756.
- ↑ 62.0 62.1 Mandal, Ananya (2009-11-26). "What is Anabolism?". News-Medical.net (in English). Retrieved 2020-07-04.
- ↑ Miziorko HM, Lorimer GH (1983). "Ribulose-1,5-bisphosphate carboxylase-oxygenase". Annual Review of Biochemistry. 52: 507–35. doi:10.1146/annurev.bi.52.070183.002451. PMID 6351728.
- ↑ Dodd AN, Borland AM, Haslam RP, Griffiths H, Maxwell K (April 2002). "Crassulacean acid metabolism: plastic, fantastic". Journal of Experimental Botany. 53 (369): 569–80. doi:10.1093/jexbot/53.369.569. PMID 11886877.
- ↑ Hügler M, Wirsen CO, Fuchs G, Taylor CD, Sievert SM (May 2005). "Evidence for autotrophic CO2 fixation via the reductive tricarboxylic acid cycle by members of the epsilon subdivision of proteobacteria". Journal of Bacteriology. 187 (9): 3020–7. doi:10.1128/JB.187.9.3020-3027.2005. PMC 1082812. PMID 15838028.
- ↑ Strauss G, Fuchs G (August 1993). "Enzymes of a novel autotrophic CO2 fixation pathway in the phototrophic bacterium Chloroflexus aurantiacus, the 3-hydroxypropionate cycle". European Journal of Biochemistry. 215 (3): 633–43. doi:10.1111/j.1432-1033.1993.tb18074.x. PMID 8354269.
- ↑ Wood HG (February 1991). "Life with CO or CO2 and H2 as a source of carbon and energy". FASEB Journal. 5 (2): 156–63. doi:10.1096/fasebj.5.2.1900793. PMID 1900793.
- ↑ Shively JM, van Keulen G, Meijer WG (1998). "Something from almost nothing: carbon dioxide fixation in chemoautotrophs". Annual Review of Microbiology. 52: 191–230. doi:10.1146/annurev.micro.52.1.191. PMID 9891798.
- ↑ Boiteux A, Hess B (June 1981). "Design of glycolysis". Philosophical Transactions of the Royal Society of London. Series B, Biological Sciences. 293 (1063): 5–22. Bibcode:1981RSPTB.293....5B. doi:10.1098/rstb.1981.0056. PMID 6115423.
- ↑ Pilkis SJ, el-Maghrabi MR, Claus TH (June 1990). "Fructose-2,6-bisphosphate in control of hepatic gluconeogenesis. From metabolites to molecular genetics". Diabetes Care. 13 (6): 582–99. doi:10.2337/diacare.13.6.582. PMID 2162755.
- ↑ 71.0 71.1 Ensign SA (July 2006). "Revisiting the glyoxylate cycle: alternate pathways for microbial acetate assimilation". Molecular Microbiology. 61 (2): 274–6. doi:10.1111/j.1365-2958.2006.05247.x. PMID 16856935.
- ↑ Finn PF, Dice JF (2006). "Proteolytic and lipolytic responses to starvation". Nutrition. 22 (7–8): 830–44. doi:10.1016/j.nut.2006.04.008. PMID 16815497.
- ↑ 73.0 73.1 Kornberg HL, Krebs HA (May 1957). "Synthesis of cell constituents from C2-units by a modified tricarboxylic acid cycle". Nature. 179 (4568): 988–91. Bibcode:1957Natur.179..988K. doi:10.1038/179988a0. PMID 13430766.
- ↑ Evans, Rhys D.; Heather, Lisa C. (June 2016). "Metabolic pathways and abnormalities". Surgery (Oxford). 34 (6): 266–272. doi:10.1016/j.mpsur.2016.03.010. ISSN 0263-9319.
- ↑ Freeze, Hudson H.; Hart, Gerald W.; Schnaar, Ronald L. (2015). "Glycosylation Precursors". In Varki, Ajit; Cummings, Richard D.; Esko, Jeffrey D. et al.. Essentials of Glycobiology (3rd ed.). Cold Spring Harbor (NY): Cold Spring Harbor Laboratory Press. http://www.ncbi.nlm.nih.gov/books/NBK453043/.
- ↑ Opdenakker G, Rudd PM, Ponting CP, Dwek RA (November 1993). "Concepts and principles of glycobiology". FASEB Journal. 7 (14): 1330–7. doi:10.1096/fasebj.7.14.8224606. PMID 8224606.
- ↑ McConville MJ, Menon AK (2000). "Recent developments in the cell biology and biochemistry of glycosylphosphatidylinositol lipids (review)". Molecular Membrane Biology. 17 (1): 1–16. doi:10.1080/096876800294443. PMID 10824734.
- ↑ Chirala SS, Wakil SJ (November 2004). "Structure and function of animal fatty acid synthase". Lipids. 39 (11): 1045–53. doi:10.1007/s11745-004-1329-9. PMID 15726818.
- ↑ White SW, Zheng J, Zhang YM (2005). "The structural biology of type II fatty acid biosynthesis". Annual Review of Biochemistry. 74: 791–831. doi:10.1146/annurev.biochem.74.082803.133524. PMID 15952903.
- ↑ Ohlrogge JB, Jaworski JG (June 1997). "Regulation of Fatty Acid Synthesis". Annual Review of Plant Physiology and Plant Molecular Biology. 48: 109–136. doi:10.1146/annurev.arplant.48.1.109. PMID 15012259.
- ↑ Dubey VS, Bhalla R, Luthra R (September 2003). "An overview of the non-mevalonate pathway for terpenoid biosynthesis in plants" (PDF). Journal of Biosciences. 28 (5): 637–46. doi:10.1007/BF02703339. PMID 14517367. Archived from the original (PDF) on 15 April 2007.
- ↑ 82.0 82.1 Kuzuyama T, Seto H (April 2003). "Diversity of the biosynthesis of the isoprene units". Natural Product Reports. 20 (2): 171–83. doi:10.1039/b109860h. PMID 12735695.
- ↑ Grochowski LL, Xu H, White RH (May 2006). "Methanocaldococcus jannaschii uses a modified mevalonate pathway for biosynthesis of isopentenyl diphosphate". Journal of Bacteriology. 188 (9): 3192–8. doi:10.1128/JB.188.9.3192-3198.2006. PMC 1447442. PMID 16621811.
- ↑ Lichtenthaler HK (June 1999). "The 1-Deoxy-D-Xylulose-5-Phosphate Pathway of Isoprenoid Biosynthesis in Plants". Annual Review of Plant Physiology and Plant Molecular Biology. 50: 47–65. doi:10.1146/annurev.arplant.50.1.47. PMID 15012203.
- ↑ 85.0 85.1 Schroepfer GJ (1981). "Sterol biosynthesis". Annual Review of Biochemistry. 50: 585–621. doi:10.1146/annurev.bi.50.070181.003101. PMID 7023367.
- ↑ Lees ND, Skaggs B, Kirsch DR, Bard M (March 1995). "Cloning of the late genes in the ergosterol biosynthetic pathway of Saccharomyces cerevisiae--a review". Lipids. 30 (3): 221–6. doi:10.1007/BF02537824. PMID 7791529.
- ↑ Himmelreich R, Hilbert H, Plagens H, Pirkl E, Li BC, Herrmann R (November 1996). "Complete sequence analysis of the genome of the bacterium Mycoplasma pneumoniae". Nucleic Acids Research. 24 (22): 4420–49. doi:10.1093/nar/24.22.4420. PMC 146264. PMID 8948633.
- ↑ Guyton, Arthur C.; Hall, John E. (2006). Textbook of Medical Physiology. Philadelphia: Elsevier. pp. 855–6. ISBN 978-0-7216-0240-0. https://archive.org/details/textbookmedicalp00acgu.
- ↑ Ibba M, Söll D (May 2001). "The renaissance of aminoacyl-tRNA synthesis". EMBO Reports. 2 (5): 382–7. doi:10.1093/embo-reports/kve095. PMC 1083889. PMID 11375928. Archived from the original on 1 May 2011.
- ↑ Lengyel P, Söll D (June 1969). "Mechanism of protein biosynthesis". Bacteriological Reviews. 33 (2): 264–301. doi:10.1128/MMBR.33.2.264-301.1969. PMC 378322. PMID 4896351.
- ↑ 91.0 91.1 Rudolph FB (January 1994). "The biochemistry and physiology of nucleotides". The Journal of Nutrition. 124 (1 Suppl): 124S–127S. doi:10.1093/jn/124.suppl_1.124S. PMID 8283301. Zrenner R, Stitt M, Sonnewald U, Boldt R (2006). "Pyrimidine and purine biosynthesis and degradation in plants". Annual Review of Plant Biology. 57: 805–36. doi:10.1146/annurev.arplant.57.032905.105421. PMID 16669783.
- ↑ Stasolla C, Katahira R, Thorpe TA, Ashihara H (November 2003). "Purine and pyrimidine nucleotide metabolism in higher plants". Journal of Plant Physiology. 160 (11): 1271–95. doi:10.1078/0176-1617-01169. PMID 14658380.
- ↑ Davies O, Mendes P, Smallbone K, Malys N (April 2012). "Characterisation of multiple substrate-specific (d)ITP/(d)XTPase and modelling of deaminated purine nucleotide metabolism" (PDF). BMB Reports. 45 (4): 259–64. doi:10.5483/BMBRep.2012.45.4.259. PMID 22531138.
- ↑ Smith JL (December 1995). "Enzymes of nucleotide synthesis". Current Opinion in Structural Biology. 5 (6): 752–7. doi:10.1016/0959-440X(95)80007-7. PMID 8749362.
- ↑ Testa B, Krämer SD (October 2006). "The biochemistry of drug metabolism--an introduction: part 1. Principles and overview". Chemistry & Biodiversity. 3 (10): 1053–101. doi:10.1002/cbdv.200690111. PMID 17193224.
- ↑ Danielson PB (December 2002). "The cytochrome P450 superfamily: biochemistry, evolution and drug metabolism in humans". Current Drug Metabolism. 3 (6): 561–97. doi:10.2174/1389200023337054. PMID 12369887.
- ↑ King CD, Rios GR, Green MD, Tephly TR (September 2000). "UDP-glucuronosyltransferases". Current Drug Metabolism. 1 (2): 143–61. doi:10.2174/1389200003339171. PMID 11465080.
- ↑ Sheehan D, Meade G, Foley VM, Dowd CA (November 2001). "Structure, function and evolution of glutathione transferases: implications for classification of non-mammalian members of an ancient enzyme superfamily". The Biochemical Journal. 360 (Pt 1): 1–16. doi:10.1042/0264-6021:3600001. PMC 1222196. PMID 11695986.
- ↑ Galvão TC, Mohn WW, de Lorenzo V (October 2005). "Exploring the microbial biodegradation and biotransformation gene pool". Trends in Biotechnology. 23 (10): 497–506. doi:10.1016/j.tibtech.2005.08.002. PMID 16125262.
- ↑ Janssen DB, Dinkla IJ, Poelarends GJ, Terpstra P (December 2005). "Bacterial degradation of xenobiotic compounds: evolution and distribution of novel enzyme activities" (PDF). Environmental Microbiology. 7 (12): 1868–82. doi:10.1111/j.1462-2920.2005.00966.x. PMID 16309386.
- ↑ Davies KJ (1995). "Oxidative stress: the paradox of aerobic life". Biochemical Society Symposium. 61: 1–31. doi:10.1042/bss0610001. PMID 8660387.
- ↑ Tu BP, Weissman JS (February 2004). "Oxidative protein folding in eukaryotes: mechanisms and consequences". The Journal of Cell Biology. 164 (3): 341–6. doi:10.1083/jcb.200311055. PMC 2172237. PMID 14757749.
- ↑ Sies H (March 1997). "Oxidative stress: oxidants and antioxidants" (PDF). Experimental Physiology. 82 (2): 291–5. doi:10.1113/expphysiol.1997.sp004024. PMID 9129943. Archived from the original (PDF) on 25 March 2009. Retrieved 9 March 2007.
- ↑ Vertuani S, Angusti A, Manfredini S (2004). "The antioxidants and pro-antioxidants network: an overview". Current Pharmaceutical Design. 10 (14): 1677–94. doi:10.2174/1381612043384655. PMID 15134565.
- ↑ von Stockar U, Liu J (August 1999). "Does microbial life always feed on negative entropy? Thermodynamic analysis of microbial growth". Biochimica et Biophysica Acta (BBA) - Bioenergetics. 1412 (3): 191–211. doi:10.1016/S0005-2728(99)00065-1. PMID 10482783.
- ↑ Demirel Y, Sandler SI (June 2002). "Thermodynamics and bioenergetics". Biophysical Chemistry. 97 (2–3): 87–111. doi:10.1016/S0301-4622(02)00069-8. PMID 12050002.
- ↑ Albert R (November 2005). "Scale-free networks in cell biology". Journal of Cell Science. 118 (Pt 21): 4947–57. arXiv:q-bio/0510054. Bibcode:2005q.bio....10054A. doi:10.1242/jcs.02714. PMID 16254242.
- ↑ Brand MD (January 1997). "Regulation analysis of energy metabolism". The Journal of Experimental Biology. 200 (Pt 2): 193–202. PMID 9050227.
- ↑ Soyer OS, Salathé M, Bonhoeffer S (January 2006). "Signal transduction networks: topology, response and biochemical processes". Journal of Theoretical Biology. 238 (2): 416–25. doi:10.1016/j.jtbi.2005.05.030. PMID 16045939.
- ↑ 110.0 110.1 Salter M, Knowles RG, Pogson CI (1994). "Metabolic control". Essays in Biochemistry. 28: 1–12. PMID 7925313.
- ↑ Westerhoff HV, Groen AK, Wanders RJ (January 1984). "Modern theories of metabolic control and their applications (review)". Bioscience Reports. 4 (1): 1–22. doi:10.1007/BF01120819. PMID 6365197.
- ↑ Fell DA, Thomas S (October 1995). "Physiological control of metabolic flux: the requirement for multisite modulation". The Biochemical Journal. 311 ( Pt 1) (Pt 1): 35–9. doi:10.1042/bj3110035. PMC 1136115. PMID 7575476.
- ↑ Hendrickson WA (November 2005). "Transduction of biochemical signals across cell membranes". Quarterly Reviews of Biophysics. 38 (4): 321–30. doi:10.1017/S0033583506004136. PMID 16600054.
- ↑ Cohen P (December 2000). "The regulation of protein function by multisite phosphorylation--a 25 year update". Trends in Biochemical Sciences. 25 (12): 596–601. doi:10.1016/S0968-0004(00)01712-6. PMID 11116185.
- ↑ Lienhard GE, Slot JW, James DE, Mueckler MM (January 1992). "How cells absorb glucose". Scientific American. 266 (1): 86–91. Bibcode:1992SciAm.266a..86L. doi:10.1038/scientificamerican0192-86. PMID 1734513.
- ↑ Roach PJ (March 2002). "Glycogen and its metabolism". Current Molecular Medicine. 2 (2): 101–20. doi:10.2174/1566524024605761. PMID 11949930.
- ↑ Newgard CB, Brady MJ, O'Doherty RM, Saltiel AR (December 2000). "Organizing glucose disposal: emerging roles of the glycogen targeting subunits of protein phosphatase-1" (PDF). Diabetes. 49 (12): 1967–77. doi:10.2337/diabetes.49.12.1967. PMID 11117996.
- ↑ Romano AH, Conway T (1996). "Evolution of carbohydrate metabolic pathways". Research in Microbiology. 147 (6–7): 448–55. doi:10.1016/0923-2508(96)83998-2. PMID 9084754.
- ↑ Koch A (1998). How did bacteria come to be?. Advances in Microbial Physiology. 40. pp. 353–99. ISBN 978-0-12-027740-7.
- ↑ Ouzounis C, Kyrpides N (July 1996). "The emergence of major cellular processes in evolution". FEBS Letters. 390 (2): 119–23. doi:10.1016/0014-5793(96)00631-X. PMID 8706840.
- ↑ Caetano-Anollés G, Kim HS, Mittenthal JE (May 2007). "The origin of modern metabolic networks inferred from phylogenomic analysis of protein architecture". Proceedings of the National Academy of Sciences of the United States of America. 104 (22): 9358–63. Bibcode:2007PNAS..104.9358C. doi:10.1073/pnas.0701214104. PMC 1890499. PMID 17517598.
- ↑ Schmidt S, Sunyaev S, Bork P, Dandekar T (June 2003). "Metabolites: a helping hand for pathway evolution?". Trends in Biochemical Sciences. 28 (6): 336–41. doi:10.1016/S0968-0004(03)00114-2. PMID 12826406.
- ↑ Light S, Kraulis P (February 2004). "Network analysis of metabolic enzyme evolution in Escherichia coli". BMC Bioinformatics. 5: 15. doi:10.1186/1471-2105-5-15. PMC 394313. PMID 15113413. Alves R, Chaleil RA, Sternberg MJ (July 2002). "Evolution of enzymes in metabolism: a network perspective". Journal of Molecular Biology. 320 (4): 751–70. doi:10.1016/S0022-2836(02)00546-6. PMID 12095253.
- ↑ Kim HS, Mittenthal JE, Caetano-Anollés G (July 2006). "MANET: tracing evolution of protein architecture in metabolic networks". BMC Bioinformatics. 7: 351. doi:10.1186/1471-2105-7-351. PMC 1559654. PMID 16854231.
- ↑ Teichmann SA, Rison SC, Thornton JM, Riley M, Gough J, Chothia C (December 2001). "Small-molecule metabolism: an enzyme mosaic". Trends in Biotechnology. 19 (12): 482–6. doi:10.1016/S0167-7799(01)01813-3. PMID 11711174.
- ↑ Spirin V, Gelfand MS, Mironov AA, Mirny LA (June 2006). "A metabolic network in the evolutionary context: multiscale structure and modularity". Proceedings of the National Academy of Sciences of the United States of America. 103 (23): 8774–9. Bibcode:2006PNAS..103.8774S. doi:10.1073/pnas.0510258103. PMC 1482654. PMID 16731630.
- ↑ Lawrence JG (December 2005). "Common themes in the genome strategies of pathogens". Current Opinion in Genetics & Development. 15 (6): 584–8. doi:10.1016/j.gde.2005.09.007. PMID 16188434. Wernegreen JJ (December 2005). "For better or worse: genomic consequences of intracellular mutualism and parasitism". Current Opinion in Genetics & Development. 15 (6): 572–83. doi:10.1016/j.gde.2005.09.013. PMID 16230003.
- ↑ Pál C, Papp B, Lercher MJ, Csermely P, Oliver SG, Hurst LD (March 2006). "Chance and necessity in the evolution of minimal metabolic networks". Nature. 440 (7084): 667–70. Bibcode:2006Natur.440..667P. doi:10.1038/nature04568. PMID 16572170.
- ↑ Rennie MJ (November 1999). "An introduction to the use of tracers in nutrition and metabolism". The Proceedings of the Nutrition Society. 58 (4): 935–44. doi:10.1017/S002966519900124X. PMID 10817161.
- ↑ Phair RD (December 1997). "Development of kinetic models in the nonlinear world of molecular cell biology". Metabolism. 46 (12): 1489–95. doi:10.1016/S0026-0495(97)90154-2. PMID 9439549.
- ↑ Sterck L, Rombauts S, Vandepoele K, Rouzé P, Van de Peer Y (April 2007). "How many genes are there in plants (... and why are they there)?". Current Opinion in Plant Biology. 10 (2): 199–203. doi:10.1016/j.pbi.2007.01.004. PMID 17289424.
- ↑ Borodina I, Nielsen J (June 2005). "From genomes to in silico cells via metabolic networks". Current Opinion in Biotechnology. 16 (3): 350–5. doi:10.1016/j.copbio.2005.04.008. PMID 15961036.
- ↑ Gianchandani EP, Brautigan DL, Papin JA (May 2006). "Systems analyses characterize integrated functions of biochemical networks". Trends in Biochemical Sciences. 31 (5): 284–91. doi:10.1016/j.tibs.2006.03.007. PMID 16616498.
- ↑ Duarte NC, Becker SA, Jamshidi N, Thiele I, Mo ML, Vo TD, et al. (February 2007). "Global reconstruction of the human metabolic network based on genomic and bibliomic data". Proceedings of the National Academy of Sciences of the United States of America. 104 (6): 1777–82. Bibcode:2007PNAS..104.1777D. doi:10.1073/pnas.0610772104. PMC 1794290. PMID 17267599.
- ↑ Goh KI, Cusick ME, Valle D, Childs B, Vidal M, Barabási AL (May 2007). "The human disease network". Proceedings of the National Academy of Sciences of the United States of America. 104 (21): 8685–90. Bibcode:2007PNAS..104.8685G. doi:10.1073/pnas.0701361104. PMC 1885563. PMID 17502601.
- ↑ Lee DS, Park J, Kay KA, Christakis NA, Oltvai ZN, Barabási AL (July 2008). "The implications of human metabolic network topology for disease comorbidity". Proceedings of the National Academy of Sciences of the United States of America. 105 (29): 9880–5. Bibcode:2008PNAS..105.9880L. doi:10.1073/pnas.0802208105. PMC 2481357. PMID 18599447.
- ↑ Csete M, Doyle J (September 2004). "Bow ties, metabolism and disease". Trends in Biotechnology. 22 (9): 446–50. doi:10.1016/j.tibtech.2004.07.007. PMID 15331224.
- ↑ Ma HW, Zeng AP (July 2003). "The connectivity structure, giant strong component and centrality of metabolic networks". Bioinformatics. 19 (11): 1423–30. CiteSeerX 10.1.1.605.8964. doi:10.1093/bioinformatics/btg177. PMID 12874056.
- ↑ Zhao J, Yu H, Luo JH, Cao ZW, Li YX (August 2006). "Hierarchical modularity of nested bow-ties in metabolic networks". BMC Bioinformatics. 7: 386. arXiv:q-bio/0605003. Bibcode:2006q.bio.....5003Z. doi:10.1186/1471-2105-7-386. PMC 1560398. PMID 16916470.
- ↑ Thykaer J, Nielsen J (January 2003). "Metabolic engineering of beta-lactam production". Metabolic Engineering. 5 (1): 56–69. doi:10.1016/S1096-7176(03)00003-X. PMID 12749845. González-Pajuelo M, Meynial-Salles I, Mendes F, Andrade JC, Vasconcelos I, Soucaille P (2005). "Metabolic engineering of Clostridium acetobutylicum for the industrial production of 1,3-propanediol from glycerol". Metabolic Engineering. 7 (5–6): 329–36. doi:10.1016/j.ymben.2005.06.001. hdl:10400.14/3388. PMID 16095939. Krämer M, Bongaerts J, Bovenberg R, Kremer S, Müller U, Orf S, et al. (October 2003). "Metabolic engineering for microbial production of shikimic acid". Metabolic Engineering. 5 (4): 277–83. doi:10.1016/j.ymben.2003.09.001. PMID 14642355.
- ↑ Koffas M, Roberge C, Lee K, Stephanopoulos G (1999). "Metabolic engineering". Annual Review of Biomedical Engineering. 1: 535–57. doi:10.1146/annurev.bioeng.1.1.535. PMID 11701499.
- ↑ "metabolism | Origin and meaning of metabolism by Online Etymology Dictionary". www.etymonline.com (in English). Retrieved 2020-07-23.
- ↑ The Lagoon: How Aristotle Invented Science. Bloomsbury. 2014. pp. 400–401. ISBN 978-1-4088-3622-4. https://archive.org/stream/lagoonhowaristot0000lero?ref=ol#page/402/mode/2up.
- ↑ Al-Roubi AS (1982). Ibn Al-Nafis as a philosopher. Symposium on Ibn al-Nafis, Second International Conference on Islamic Medicine. Kuwait: Islamic Medical Organization. (cf. Ibn al-Nafis As a Philosopher, Encyclopedia of Islamic World [1])
- ↑ Eknoyan G (1999). "Santorio Sanctorius (1561-1636) - founding father of metabolic balance studies". American Journal of Nephrology. 19 (2): 226–33. doi:10.1159/000013455. PMID 10213823.
- ↑ Modern Development of the Chemical and Biological Sciences. A History of Science: in Five Volumes. IV. New York: Harper and Brothers. 1904. pp. 184–185. https://archive.org/details/historyofscience04willuoft/page/n7/mode/2up.
- ↑ Manchester KL (December 1995). "Louis Pasteur (1822-1895)--chance and the prepared mind". Trends in Biotechnology. 13 (12): 511–5. doi:10.1016/S0167-7799(00)89014-9. PMID 8595136.
- ↑ Kinne-Saffran E, Kinne RK (1999). "Vitalism and synthesis of urea. From Friedrich Wöhler to Hans A. Krebs". American Journal of Nephrology. 19 (2): 290–4. doi:10.1159/000013463. PMID 10213830.
- ↑ Eduard Buchner's 1907 Nobel lecture at http://nobelprize.org Accessed 20 March 2007
- ↑ Kornberg H (December 2000). "Krebs and his trinity of cycles". Nature Reviews. Molecular Cell Biology. 1 (3): 225–8. doi:10.1038/35043073. PMID 11252898.
- ↑ Krebs HA, Henseleit K (1932). "Untersuchungen über die Harnstoffbildung im tierkorper". Z. Physiol. Chem. 210 (1–2): 33–66. doi:10.1515/bchm2.1932.210.1-2.33.
Krebs HA, Johnson WA (April 1937). "Metabolism of ketonic acids in animal tissues". The Biochemical Journal. 31 (4): 645–60. doi:10.1042/bj0310645. PMC 1266984. PMID 16746382.
更多资料
- The Chemistry of Life.. Penguin Press Science. 1999. ISBN 0-14-027273-9.
- Into the Cool: Energy Flow, Thermodynamics, and Life.. University of Chicago Press. 2005. ISBN 0-226-73936-8.
- Oxygen: The Molecule that Made the World.. USA: Oxford University Press. 2004. ISBN 0-19-860783-0.
- Fundamentals of Enzymology: Cell and Molecular Biology of Catalytic Proteins.. Oxford University Press. 1999. ISBN 0-19-850229-X.
- Biochemistry. W. H. Freeman and Company. 2002. ISBN 0-7167-4955-6.
- Lehninger Principles of Biochemistry.. Palgrave Macmillan. 2004. ISBN 0-7167-4339-6.
- Brock's Biology of Microorganisms.. Benjamin Cummings. 2002. ISBN 0-13-066271-2.
- The Biological Chemistry of the Elements: The Inorganic Chemistry of Life.. Clarendon Press. 1991. ISBN 0-19-855598-9.
- Bioenergetics. Academic Press Inc.. 2002. ISBN 0-12-518121-3.
- Wood HG (February 1991). "Life with CO or CO2 and H2 as a source of carbon and energy". FASEB Journal. 5 (2): 156–63. doi:10.1096/fasebj.5.2.1900793. PMID 1900793.
外部链接
General information
- The Biochemistry of Metabolism
- Sparknotes SAT biochemistry Overview of biochemistry. School level.
- MIT Biology Hypertextbook Undergraduate-level guide to molecular biology.
Human metabolism
- Topics in Medical Biochemistry Guide to human metabolic pathways. School level.
- THE Medical Biochemistry Page Comprehensive resource on human metabolism.
Databases
- Flow Chart of Metabolic Pathways at ExPASy
- IUBMB-Nicholson Metabolic Pathways Chart
- SuperCYP: Database for Drug-Cytochrome-Metabolism
Metabolic pathways
编者推荐
集智文章
吉布斯——热力学大师与统计物理奠基人
本文介绍被爱因斯坦称之为“美国历史上最杰出的英才”的吉布斯的生平事迹。他一生致力于物理光学、热力学以及后来他首创的统计力学,硕果累累的他对于学术界来说无疑是一个巨大的财富。但同时,吉布斯是一个私下喜欢带学生去爬山的老师。他认为登山者与物理学家的相似之处都是在艰难的攀登中自得其乐。
集智视频
热力学第二定律与耗散结构
本课程难度为中级。讲师为张江,北京师范大学系统科学学院教授、博士生导师,集智俱乐部创始人,集智学园创始人。研究方向包括复杂网络与机器学习、复杂系统分析与建模、计算社会科学。目前主要的研究课题是复杂系统自动建模。
本课程中,将带领大家认识热力学第二定律,重点关注熵是什么,以及它为什么可以构成万事万物演化的时间之箭。
爱因斯坦说过,你可以不知道牛顿第二定律,但你不能不知道热力学第二定律。熵到底是什么?它为什么可以构成万事万物演化的时间之箭?令人意想不到的是,我们不仅仅是一个虚无缥缈的斑图,更是一个处于流变之中的耗散结构斑图。你只有不断开放,从系统获取负熵,才能勉强维持生存。
复杂性研究的生物学视角|生命复杂性系列读书会第三期
本课程难度为中级。讲师为小木球(肖沐秋),肿瘤分子遗传学博士,集智科学家,从事生物新药研发工作。曾在集智俱乐部组织过24期《失控》读书会,长期关注生命复杂性和生命起源问题。
生命是什么?生命怎样起源?生命怎样演化?这些是对生命现象的本质追问,除了传统的生物学研究,如今有大量来自信息、物理、计算机领域的工具方法,正在揭开生命复杂性谜题。
本期分享,将从生物学视角,梳理复杂性研究历程、工具、方法以及误区,并从模式生成和主体生成两个方向探讨系统生物学及生命起源问题。
本中文词条由Solitude编译,Fernando审校,糖糖编辑,如有问题,欢迎在讨论页面留言。
本词条内容源自wikipedia及公开资料,遵守 CC3.0协议。