“XNA”的版本间的差异
| 第15行: | 第15行: | ||
| − | 在特殊的聚合酶被开发出来之前,对XNA的研究并不多,这种酶能够以DNA为模板复制出XNA,也能将XNA复制回DNA。<ref name=":1">{{Cite web |last=Gonzales |first=Robbie | + | 在特殊的聚合酶被开发出来之前,对XNA的研究并不多,这种酶能够以DNA为模板复制出XNA,也能将XNA复制回DNA。<ref name=":1">{{Cite web |last=Gonzales |first=Robbie |title=XNA Is Synthetic DNA That's Stronger than the Real Thing |website=[[Io9]] |date=19 April 2012 |access-date=15 October 2015 |url=http://io9.com/5903221/meet-xna-the-first-synthetic-dna-that-evolves-like-the-real-thing}}</ref>例如Pinheiro等人在2012年的一项研究已证明了一种可以在大约100bp序列长度上工作的XNA聚合酶。<ref name="Synthetic genetic polymers capable">{{cite journal | vauthors = Pinheiro VB, Taylor AI, Cozens C, Abramov M, Renders M, Zhang S, Chaput JC, Wengel J, Peak-Chew SY, McLaughlin SH, Herdewijn P, Holliger P | display-authors = 6 | title = Synthetic genetic polymers capable of heredity and evolution | journal = Science | volume = 336 | issue = 6079 | pages = 341–344 | date = April 2012 | pmid = 22517858 | pmc = 3362463 | doi = 10.1126/science.1217622 | bibcode = 2012Sci...336..341P }}</ref>最近,合成生物学家菲利普·霍利格 Philipp Holliger和亚历山大·泰勒 Alexander Taylor成功地创造了XNAzymes。XNAzymes相当于核酶,由DNA或核糖核酸构成。这表明,这表明XNAs不仅存储遗传信息,还可以作为酶,这提高了其他地方的生命可能起源于RNA或DNA以外物质的可能性。<ref>{{Cite web |website=[[Medical Research Council (United Kingdom)|Medical Research Council]] |title=World's first artificial enzymes created using synthetic biology |date=1 December 2014 |url=http://www.mrc.ac.uk/news/browse/world-s-first-artificial-enzymes-created-using-synthetic-biology/}}</ref> |
| 第36行: | 第36行: | ||
| − | HNA可被用作识别和结合特定序列的药物。科学家已经能够分离出HNAs,以靶向结合艾滋病毒相关的基因序列。<ref>{{Cite web |title = Polymers perform non-DNA evolution |url = http://www.rsc.org/chemistryworld/News/2012/April/xna-hna-primordial-life-dna-evolution-chemistry.asp |first=Andy |last=Extance | + | HNA可被用作识别和结合特定序列的药物。科学家已经能够分离出HNAs,以靶向结合艾滋病毒相关的基因序列。<ref>{{Cite web |title = Polymers perform non-DNA evolution |url = http://www.rsc.org/chemistryworld/News/2012/April/xna-hna-primordial-life-dna-evolution-chemistry.asp |first=Andy |last=Extance |date=19 April 2012 |website = [[Royal Society of Chemistry]] |access-date = 15 October 2015}}</ref>研究表明,在存在环己烯核酸的情况下,具有类似D型立体化学结构的CeNAs可以与自身和RNA形成稳定的双链体。结果表明,当CeNAs与DNA形成双链体时,CeNAs不是很稳定。<ref>{{cite journal | vauthors = Gu P, Schepers G, Rozenski J, Van Aerschot A, Herdewijn P | title = Base pairing properties of D- and L-cyclohexene nucleic acids (CeNA) | journal = Oligonucleotides | volume = 13 | issue = 6 | pages = 479–489 | year = 2003 | pmid = 15025914 | doi = 10.1089/154545703322860799 }}</ref> |
==启示== | ==启示== | ||
2022年4月17日 (日) 09:46的版本
异源核酸 Xeno nucleic acids(XNA)是人工合成的核酸类似物,具有不同于天然核酸DNA和RNA的糖骨架。[1]截止至2011年,已证明至少有六种合成的糖类能形成可存储和检索基因信息的核酸骨架。目前正在研究用合成的聚合酶来转化XNA。对其生产和应用的研究创造了一个被称为异源生物学 xenobiology的领域。
虽然遗传信息仍然存储在四个标准碱基对(不像其他核酸类似物那样),但是天然的DNA聚合酶不能读取和复制这些信息。因此,存储在XNA中的遗传信息是“不可视的”,对于以天然DNA为基础的生物体来说是无用的。[2]
背景
DNA的结构于1953年被发现。大约在21世纪初,研究人员创造了许多奇特的类DNA结构,XNA。XNA是一种人工合成的聚合物,可以携带与DNA相同的信息,但具有不同的分子成分。XNA中的“X”代表“ xeno”,意思是外乡人或外星人,以表示其分子结构与DNA或RNA相比的差异。[3]
在特殊的聚合酶被开发出来之前,对XNA的研究并不多,这种酶能够以DNA为模板复制出XNA,也能将XNA复制回DNA。[3]例如Pinheiro等人在2012年的一项研究已证明了一种可以在大约100bp序列长度上工作的XNA聚合酶。[4]最近,合成生物学家菲利普·霍利格 Philipp Holliger和亚历山大·泰勒 Alexander Taylor成功地创造了XNAzymes。XNAzymes相当于核酶,由DNA或核糖核酸构成。这表明,这表明XNAs不仅存储遗传信息,还可以作为酶,这提高了其他地方的生命可能起源于RNA或DNA以外物质的可能性。[5]
Structure 结构
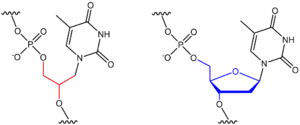
这张图片显示了XNAs中使用的糖骨架与生物学中常见的DNA和RNA之间的差异。DNA链和RNA链是由名为核苷酸的长链分子串联而成的。核苷酸由三种化学成分组成:磷酸、五碳糖(可以是脱氧核糖,它给DNA提供了“D”,也可以是核糖,即RNA中的“R”) 、以及五种标准碱基之一(腺嘌呤 adenine、鸟嘌呤 guanine、胞嘧啶 cytosine、胸腺嘧啶 thymine或尿嘧啶 uracil)。
组成这六种异源核酸的分子与DNA和RNA的分子几乎完全相同,只有一处例外:在XNA核苷酸中,DNA和RNA的脱氧核糖和核糖被其他化学结构所取代。这些替换使得XNAs功能和结构上与DNA和RNA相似,尽管它们是非自然的、人工的。
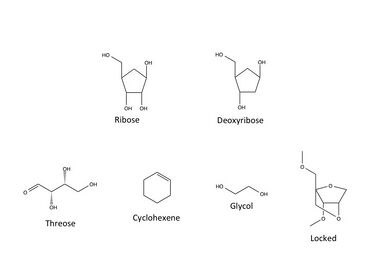
XNA相对于DNA和RNA能表现出各种结构上的化学变化。迄今为止合成的XNA类型包括: [2]
- 1,5-脱水己醇核酸(HNA)
- 环己烯核酸(CeNA)
- 苏糖核酸(TNA)
- 乙二醇核酸(GNA)
- 锁核酸(LNA)
- 肽核酸(PNA)
- 氟阿拉伯核酸(FANA)
HNA可被用作识别和结合特定序列的药物。科学家已经能够分离出HNAs,以靶向结合艾滋病毒相关的基因序列。[6]研究表明,在存在环己烯核酸的情况下,具有类似D型立体化学结构的CeNAs可以与自身和RNA形成稳定的双链体。结果表明,当CeNAs与DNA形成双链体时,CeNAs不是很稳定。[7]
启示
XNA研究的目的不是为了让科学家更好地了解历史上发生过的生物进化,而是探索我们能够控制甚至重新编程生物体基因组成的方法。XNA在解决目前转基因生物的遗传污染问题上显示出巨大潜力。[8]尽管DNA在储存遗传信息和提供复杂生物多样性方面具有令人难以置信的效率,但其四个字母的遗传密码表相对有限。使用6个XNAs的遗传密码而不是4个自然产生的DNA碱基,或将展现出基因修饰和扩展化学功能的无尽机遇。[9]
关于XNAs的各种假说和理论的发展已经改变了目前我们理解核酸的一个关键点: 遗传和进化并不是人们曾经认为的那样只局限于DNA和RNA,而仅仅是一种起源于能够储存信息的聚合物的简单过程。对于XNAs的研究将允许研究人员评估DNA和RNA是不是最有效和最理想的生命构件,或者这两种分子是不是在更大类的化学祖先进化之后被随机地选择出来的。[10]
应用
一种利用XNA的理论是将其作为抗病剂纳入医学。目前,一些用于各种疾病治疗的酶和抗体在胃或血液中分解得太快。但由于XNA是外来的,而且人类尚未进化出能够分解它们的酶,因此对于目前正在使用的DNA和RNA治疗方法,XNA可能成为比它们更持久的替换物。[11]
XNA实验已经可以替换和扩大遗传密码表,而且XNA已经显示出与DNA和RNA核苷酸的互补性,这表明它具有转录和重组的潜力。在佛罗里达大学进行的一项实验中,通过AEISIS-SELEX(人工扩增的遗传信息系统——通过指数富集的配体的系统进化)产生了一种XNA适配体,并成功与乳腺癌细胞系结合。[12]此外,在大肠杆菌中的实验表明,XNA在体内具有作为DNA生物模板的能力。[13]
在推进对XNAs的基因研究时,必须考虑到有关生物安全、生物防护、伦理和治理/监管的各种问题。[2]这里的一个关键问题是,在体内环境中,XNA是否会与自然环境中的DNA和RNA混合,从而使科学家无法控制或预测其在基因突变中可能的影响。[11]
XNA还可以作催化剂,就像RNA可以当作酶一样。研究人员已经证明,XNA能够剪切和连接DNA、RNA和其他XNA序列,其中XNA对XNA分子的催化反应最具活性。这项研究可以用来确定DNA和RNA在生命中的作用是通过自然选择过程产生的,还是说仅仅是一个巧合。[14]
XNA可以通过与靶DNA序列杂交,作为定量实时聚合酶链式反应 quantitative real-time polymerase chain reaction(qPCR)的分子钳。[15]在《公共科学图书馆》(PLOS ONE)期刊上发表的一项研究中,一种DNA介导的分子钳技术检测到了癌前结直肠癌 colorectal cancer(CRC)病变和结直肠癌的突变无细胞DNA cell-free DN(cfDNA)。[15]XNA还可以作为检测核酸靶序列的高特异性分子探针。[16]
参见
参考文献
- ↑ Schmidt, Markus (2012). Synthetic Biology. John Wiley & Sons. pp. 151–. ISBN 978-3-527-65926-5. https://books.google.com/books?id=lr0SgX1Hx2gC&pg=PA151.
- ↑ 2.0 2.1 2.2 Schmidt M (April 2010). "Xenobiology: a new form of life as the ultimate biosafety tool". BioEssays. 32 (4): 322–331. doi:10.1002/bies.200900147. PMC 2909387. PMID 20217844.
- ↑ 3.0 3.1 Gonzales, Robbie (19 April 2012). "XNA Is Synthetic DNA That's Stronger than the Real Thing". Io9. Retrieved 15 October 2015.
- ↑ Pinheiro VB, Taylor AI, Cozens C, Abramov M, Renders M, Zhang S, et al. (April 2012). "Synthetic genetic polymers capable of heredity and evolution". Science. 336 (6079): 341–344. Bibcode:2012Sci...336..341P. doi:10.1126/science.1217622. PMC 3362463. PMID 22517858.
- ↑ "World's first artificial enzymes created using synthetic biology". Medical Research Council. 1 December 2014.
- ↑ Extance, Andy (19 April 2012). "Polymers perform non-DNA evolution". Royal Society of Chemistry. Retrieved 15 October 2015.
- ↑ Gu P, Schepers G, Rozenski J, Van Aerschot A, Herdewijn P (2003). "Base pairing properties of D- and L-cyclohexene nucleic acids (CeNA)". Oligonucleotides. 13 (6): 479–489. doi:10.1089/154545703322860799. PMID 15025914.
- ↑ Herdewijn P, Marlière P (June 2009). "Toward safe genetically modified organisms through the chemical diversification of nucleic acids". Chemistry & Biodiversity. 6 (6): 791–808. doi:10.1002/cbdv.200900083. PMID 19554563.
- ↑ Pinheiro VB, Holliger P (August 2012). "The XNA world: progress towards replication and evolution of synthetic genetic polymers". Current Opinion in Chemical Biology. 16 (3–4): 245–252. doi:10.1016/j.cbpa.2012.05.198. PMID 22704981.
- ↑ Hunter P (May 2013). "XNA marks the spot. What can we learn about the origins of life and the treatment of disease through artificial nucleic acids?". EMBO Reports. 14 (5): 410–413. doi:10.1038/embor.2013.42. PMC 3642382. PMID 23579343.
- ↑ 11.0 11.1 "XNA: Synthetic DNA That Can Evolve". Popular Mechanics. 19 April 2012. Retrieved 17 November 2015.
- ↑ Sefah K, Yang Z, Bradley KM, Hoshika S, Jiménez E, Zhang L, et al. (January 2014). "In vitro selection with artificial expanded genetic information systems". Proceedings of the National Academy of Sciences of the United States of America. 111 (4): 1449–1454. Bibcode:2014PNAS..111.1449S. doi:10.1073/pnas.1311778111. PMC 3910645. PMID 24379378.
- ↑ Pezo V, Liu FW, Abramov M, Froeyen M, Herdewijn P, Marlière P (July 2013). "Binary genetic cassettes for selecting XNA-templated DNA synthesis in vivo". Angewandte Chemie. 52 (31): 8139–8143. doi:10.1002/anie.201303288. PMID 23804524.
- ↑ Taylor AI, Pinheiro VB, Smola MJ, Morgunov AS, Peak-Chew S, Cozens C, et al. (February 2015). "Catalysts from synthetic genetic polymers". Nature. 518 (7539): 427–430. Bibcode:2015Natur.518..427T. doi:10.1038/nature13982. PMC 4336857. PMID 25470036.
- ↑ 15.0 15.1 Sun Q, Pastor L, Du J, Powell MJ, Zhang A, Bodmer W, et al. (2021-10-05). "A novel xenonucleic acid-mediated molecular clamping technology for early colorectal cancer screening". PLOS ONE. 16 (10): e0244332. Bibcode:2021PLoSO..1644332S. doi:10.1371/journal.pone.0244332. PMC 8491914. PMID 34610014.
- ↑ D'Agata R, Giuffrida MC, Spoto G (November 2017). "Peptide Nucleic Acid-Based Biosensors for Cancer Diagnosis". Molecules. 22 (11): 1951. doi:10.3390/molecules22111951. PMC 6150339. PMID 29137122.
编者推荐
集智课程
[]
本中文词条由余凡尘审校,薄荷编辑,如有问题,欢迎在讨论页面留言。
本词条内容源自wikipedia及公开资料,遵守 CC3.0协议。