“AP算法”的版本间的差异
| 第35行: | 第35行: | ||
[[File:affinitypropagation_figure_5.png|center|400px]] | [[File:affinitypropagation_figure_5.png|center|400px]] | ||
| − | + | 因为责任和可用性两种信息是在相似性矩阵的基础上计算和迭代,所以算法只利用了相似性信息。当矩阵中有大量元素为空的时候,AP仍然可以给出聚类,而且速度更快。 | |
==劣势== | ==劣势== | ||
2020年5月27日 (三) 17:35的版本
简介
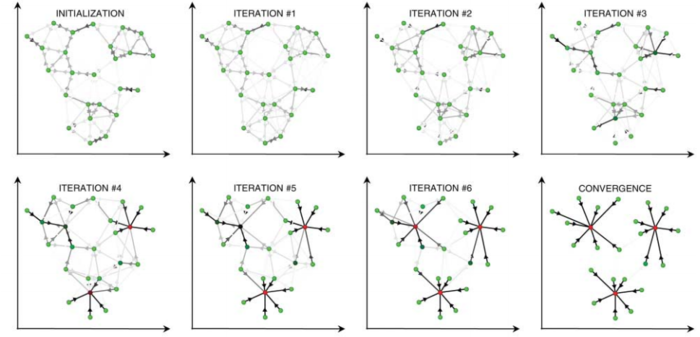
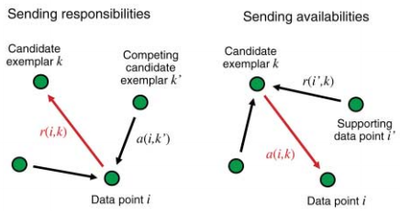
affinity propagation是Frey和Dueck在2007年发表在Science上的聚类算法。从更广义的角度说,它属于消息传递算法的一种。原始文献见这里,这里有关于AP算法的问答,包括适用的数据量大小和计算速度等细节。和k-means等它聚类算法一样,它的输入是一个N * N 的相似矩阵。在这个相似矩阵上,算法通过在数据点之间传递信息(责任和可用性,前者决定点i有多大意愿选择k作为自己的代表例子,后者决定k有多大意愿决定把i选择做代表),不断修改聚类中心的数量和位置,直到整个数据的净相似性(聚类中心k自己对自己的相似性+所有节点i!=k到k的相似性)达到最大。
Marc Mézard在同期Science对Frey和Dueck的文章给出了一个有趣的评论,他应用了卡拉瓦乔的一幅画,通过人物(数据点)之间的手势和眼神的交流来解释AP是如何工作,选出一群人的焦点的(见下图)。
优势
比起k-means方法,这种方法的好处是:
不需要事先指定聚类的数量
在affinity propagation中聚类的数量是由偏好的设定和数据的结构共同决定的。一般默认将所有的数据点的偏好设为相似矩阵的中位数。节点会比较自己的偏好和与其他节点之间的相似性,如果后者更高,就选择后者作为例子,反之则自己做例子。所以一开始如果把所有的偏好都设成0或大于0,而相似矩阵上的元素是负值(例如负的欧式距离),那么每个节点都会成为例子,也就是最后举出来N类。如果偏好被设成一个比较小的值,例如相似矩阵的中位数,那么就会在迭代过程中自发形成聚类的团块数量。
聚类的结果不会变化
kmeans方法是先随机抽样生成聚类中心或者例子,但AP则是在每一轮中同时考虑所有的节点,所以后者是一个确定性模型,也就意味着聚类的结果不会随机变化。
适用于非对称相似性矩阵,甚至不满足三角关系的相似性数据
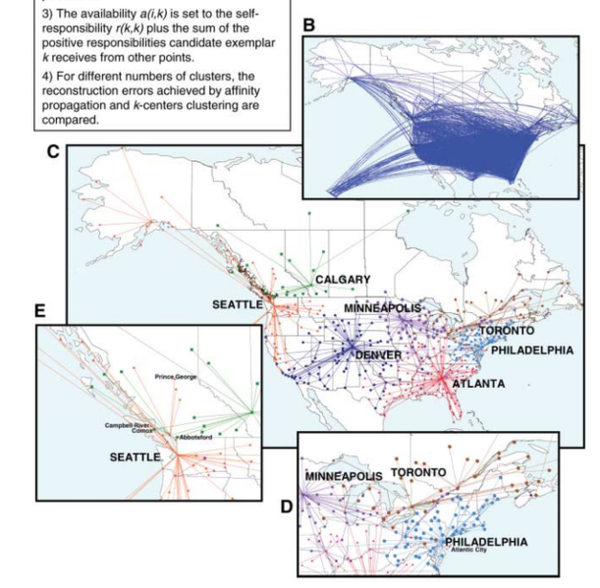
kmeans方法中,相似性矩阵是对称的,即s(i,j)=s(j,i),但是AP并没有这个要求。后者甚至适用于不满足三角关系的情况。在2007年的论文中,作者分析的美国和加拿大的航班网络(网络也可以看做是一个相似性矩阵,相似性权重为网络连边上的流量或者别的统计量,本案例中是一个城市到另一个城市的飞行时间)。在这个网络里,相似性是城市之间的飞行时间,这个变量是不满足三角关系的。例如A->B的平均飞行时间不一定就小于A->C+C->B的平均飞行时间(因为前者可能会没有直达飞机而要中途在别处转机,而且地球是一个球体)。在这个案例中,AP给出了很好的划分结果。
适用于稀疏的相似性矩阵
因为责任和可用性两种信息是在相似性矩阵的基础上计算和迭代,所以算法只利用了相似性信息。当矩阵中有大量元素为空的时候,AP仍然可以给出聚类,而且速度更快。
劣势
AP的劣势是充分识别了数据内部的结构后,对于许多真实数据,都会聚出比较多的类,因此在聚类数量在1-5时,速度没有K-means快,效果也不一定好。 具体来说,AP算法复杂度为O(N*N*logN),而K-Means是O(N*K)。因此当N比较大时(N>3000),AP聚类算法往往需要算很久。
应用
因为AP只是一种聚类算法,所以在机器学习里是non domain-specific的,文章中给出了四个例子,对生物基因的聚类,对人脸的聚类,对文章主题句的提取(这个比较有意思,作者把自己发表的这篇文章提出四句话),和对城市之间航运网络的聚类。
在python的sklearn包里带了AP算法,应用起来也是很方便的:
首先调用包
from sklearn.cluster import AffinityPropagation
from sklearn import metrics
from sklearn.datasets.samples_generator import make_blobs
import pylab as pl
from itertools import cycle
其次生成模拟数据
# Generate sample data
centers = [[1, 1], [-1, -1], [1, -1]]
X, labels_true = make_blobs(n_samples=300, centers=centers, cluster_std=0.5,
random_state=0)
接着拟合AP
# Compute Affinity Propagation
af = AffinityPropagation(preference=-50).fit(X)
cluster_centers_indices = af.cluster_centers_indices_
labels = af.labels_
n_clusters_ = len(cluster_centers_indices)
print('Estimated number of clusters: %d' % n_clusters_)
print("Homogeneity: %0.3f" % metrics.homogeneity_score(labels_true, labels))
print("Completeness: %0.3f" % metrics.completeness_score(labels_true, labels))
print("V-measure: %0.3f" % metrics.v_measure_score(labels_true, labels))
print("Adjusted Rand Index: %0.3f"
% metrics.adjusted_rand_score(labels_true, labels))
print("Adjusted Mutual Information: %0.3f"
% metrics.adjusted_mutual_info_score(labels_true, labels))
print("Silhouette Coefficient: %0.3f"
% metrics.silhouette_score(X, labels, metric='sqeuclidean'))
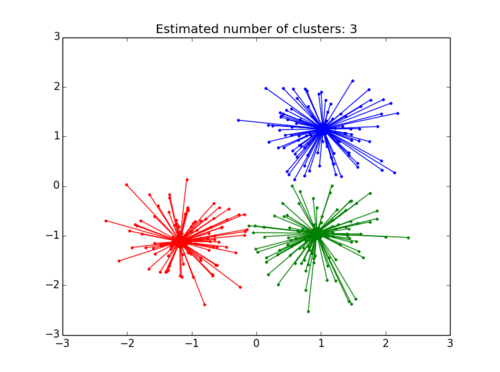
最后绘制出结果
# Plot result
pl.close('all')
pl.figure(1)
pl.clf()
colors = cycle('bgrcmykbgrcmykbgrcmykbgrcmyk')
pl.title('Estimated number of clusters: %d' % n_clusters_)
for k, col in zip(range(n_clusters_), colors):
class_members = labels == k
cluster_center = X[cluster_centers_indices[k]]
pl.plot(X[class_members, 0], X[class_members, 1], col + '.')
pl.plot(cluster_center[0], cluster_center[1], 'o', markerfacecolor=col,
markeredgecolor='k', markersize=14)
for x in X[class_members]:
pl.plot([cluster_center[0], x[0]], [cluster_center[1], x[1]], col)
每次运行的结果都是一样的。