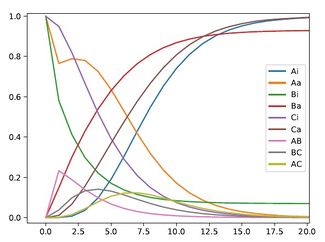
系统生物学
系统生物学 Systems biology是对复杂生物系统进行演算分析、数学分析和建模的学科。它是一个以生物学为基础的跨学科研究领域,侧重于生物系统内复杂的相互作用,采用整体的方法(整体论 holism而不是更传统的还原论 reductionism)进行生物学研究。[1]它跨越了系统论和应用数学方法的领域,发展成为复杂系统生物学 complex systems biology的一个分支。
特别是从2000年起,这个概念在生物学中被广泛应用于各种场合。人类基因组计划 Human Genome Project是生物学中应用系统思维的一个例子,它在遗传学这个生物学领域中引入了新的协作型工作方式。
系统生物学的目标之一是模拟和发现细胞 cells、组织 tissues和有机体 organisms作为一个系统运作的涌现特性,其理论描述只有使用系统生物学技术才有可能实现。[2][1]
概述
系统生物学可以从许多不同的方面来考虑。
系统生物学作为一个研究领域,具体探讨关于生物系统的组成部分之间的互动,以及各系统要素的相互作用如何产生该系统的功能和行为(例如,代谢通路中或心跳时产生的酶和代谢物)。
作为一种研究范式,系统生物学通常被认为与所谓的还原论范式(生物组织)相对立,尽管它完全符合科学方法。以下几句话中提到了两种范式之间的区别: “还原论方法成功地确定了大多数组成部分和许多相互作用,但不幸的是,没有提供令人信服的概念或方法来理解系统特性是如何出现的... 通过多个组分同时进行定量测量”改为“通过观察多个分组同时进行的定量实验。”(Sauer等人)“系统生物学...是合并而不是分解,是整合而不是简化。它要求我们建立起与我们的还原论方法一样严谨但不同的整合思维方式...这意味着彻底改变我们的哲学。”(丹尼斯·诺贝尔 Dennis Nobel)
作为一系列用于进行研究的操作方案,即一个由理论、分析或计算模型组成的循环,系统生物学提出关于生物系统的具体可检验的假设,接着进行实验验证,然后使用新获得的细胞或细胞过程的定量描述来优化计算模型或理论。由于目标是一个系统中相互作用的模型,所以最适合系统生物学的实验技术就是那些全系统范围的、尽可能完整的实验技术。因此,转录组学、代谢组学、蛋白质组学和高通量技术被用来收集定量数据,从而用于模型的建立和验证。
作为动力系统理论在分子生物学领域的应用,系统生物学对所研究的系统在动力学上的关注正是它和生物信息学之间的主要概念差异。[3]
作为一种社会科学现象,系统生物学由利用多样的跨学科的工具和人员的实验资源,寻求整合有关生物系统相互作用的复杂数据的战略所定义。[4]
各种各样的观点说明了这样一个事实,即系统生物学指的是一系列周边重叠概念的集合,而不是一个独立的领域。然而,随着系统生物学的教职和研究机构在全球范围内的激增,这个术语在2007年已经广泛流行和普及。
历史
系统生物学根植于酶动力学 enzyme kinetics的定量模型(酶动力学在1900年到1970年间蓬勃发展)、种群动力学 population dynamics的数学模型、神经生理学模拟、控制理论和控制论 control theory以及协同学 Synergetics。
理论家路德维希·冯·贝塔郎菲 Ludwig von Bertalanffy和他的一般系统论 general systems theory可以被看作是系统生物学先驱之一。英国神经生理学家、诺贝尔奖获得者艾伦·劳埃德·霍奇金 Alan Lloyd Hodgkin和安德鲁·费尔丁·赫克斯利 Andrew Ferdinand Hoxley在1952年发表了最早的细胞生物学的数理分析之一,他们也创建了一个数学模型,解释了沿神经元细胞轴突传播的动作电位。他们的模型描述了一种由钾和钠两种不同的分子成分之间的相互作用所产生的细胞功能,所以这可以被看作是演算系统生物学的开端。
无独有偶,艾伦·图灵 Alan Turing在1952年发表了形态发生的化学基础,描述了最初同质的生物系统中是如何产生不均匀性的。[5]
1960年,Dennis Noble创建了第一个心脏起搏器的计算模型。
系统理论家米哈伊洛·梅萨罗维奇 Mikhailo Mesarovich于1966年在俄亥俄州克利夫兰市的凯斯理工学院召开了一次题为“系统理论与生物学”的国际研讨会,开启了系统生物学作为一个独特领域的正式研究。
人们在20世纪六七十年代研究出几种研究复杂分子系统的方法,如代谢控制分析和生化系统理论。
整个20世纪80年代分子生物学的成功,以及人们对理论生物学的怀疑,加之收获小于预期,使得生物过程的定量模拟成为一个日渐被轻视的领域。
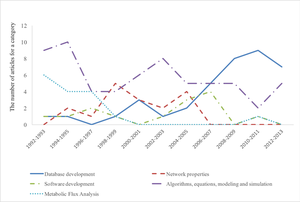
然而,20世纪90年代功能基因组学的诞生意味着人们可以得到大量高质量的数据,同时计算能力爆炸式增长,使得更真实的模型成为可能。1992年至1994年,曾斌撰写系列关于系统医学、系统遗传学和系统生物工程方面的文章[7][8][9][10][11][12],1996年,他在北京举行的第一届国际转基因动物会议上作了关于生物系统理论和系统方法研究的演讲。1997年,富田正丸小组发表了第一个关于整个(假设的)细胞新陈代谢的定量模型。
2000年左右,在西雅图和东京建立了系统生物学研究所之后,由于各种基因组项目的完成、组学(如基因组学和蛋白质组学)数据的大量增加以及随之而来的高通量实验和生物信息学的进展,系统生物学作为一项独立的运动而涌现出来。不久之后,第一个完全致力于系统生物学的院系成立了(例如,哈佛医学院的系统生物学系)[13]。
2003年,麻省理工学院的研究从CytoSolve开始,这是一种通过动态整合多个分子通路模型来建立整个细胞模型的方法。[14]从那时起,各种致力于系统生物学的研究机构已经发展起来。例如,美国国立卫生研究院的 NIGMS 建立了一个项目补助金,目前正在支持美国的十多个系统生物学中心。[15]截至2006年夏天,由于系统生物学人才短缺,全球多地建起了系统生物学博士培养计划。
同年,美国国家科学基金会 National Science Foundation(NSF)提出了二十一世纪系统生物学的一个巨大挑战:为整个细胞建立数学模型。
2012年,纽约西奈山伊坎医学院的卡尔实验室完成了第一个对整个生殖支原体细胞的建模。该细胞模型能够预测基因变异后的生殖支原体细胞的存活时间。[16]
系统生物学发展的一个重要里程碑是系统生物学变成了一个国际性课题。
相关领域
系统生物学具有利用跨学科工具从多个实验来源获取、整合和分析复杂数据集的能力,一些典型的技术平台包括:
- 表型组学,即生物表型在其生命周期内的变化。基因组学,即生物脱氧核糖核酸序列、包括生物内部细胞特异性变异(例如端粒长度变化);
- 表观基因组学或表观遗传学,生命体和相应的细胞特异性转录调控因子没有经验性地编码在基因组序列中(例如 DNA 甲基化、组蛋白乙酰化和脱乙酰化等);
- 转录组学,通过 DNA 微阵列或基因表达的系列分析来测量生物体、组织或整个细胞的基因表达。干扰素组学,即生物体、组织或细胞水平的转录校正因子(例如RNA干扰);
- 蛋白质组学,通过二维凝胶电泳、质谱法或多维蛋白质识别技术(先进的高效液相色谱系统加上质谱法),进行生物体、组织或细胞水平的蛋白质和多肽测量。子学科包括磷酸蛋白质组学、糖蛋白质组学和其他检测化学修饰蛋白质的方法。
- 代谢组学,测量有机体、细胞或组织水平系统中被称为代谢物的小分子;
- 糖组学,有机体、组织或细胞水平的碳水化合物测量;
- 脂质组学,有机体、组织或细胞水平的脂质测量。[17]
除了识别和定量化上述给定的分子之外,有进一步的技术来分析细胞内的动力学和相互作用。研究的相互作用包括生物、组织、细胞和细胞内分子的相互作用(相互作用组学)。[18]目前,在这一领域的权威分子学科,尽管这一效用的定义并不仅仅局限于该领域,也有其它分子学科的作用。这些分子学科包括: 神经电动力学,这是一个有机体网络,其中大脑的计算功能作为一个动态系统,包括潜在的生物物理机制和新兴的电力相互作用的计算[19];流体学,测量一个系统里分子随着时间的动态变化,如细胞、组织或有机体;[20]在处理系统生物学问题时,有两种主要的方法。它们分别是自上而下和自下而上的方法。自上而下的方法尽可能多把系统考虑在内,并且在很大程度上依赖于实验结果。RNA-seq 技术是自上而下实验方法的一个例子。相反,自下而上的方法用于创建详细的模型,同时也结合了实验数据。自下而上方法的一个例子是使用电路模型来描述一个简单的基因网络。[21]
有各种技术用于捕获mRNA、蛋白质的动态变化和翻译后修饰。如生物力学 Mechanobiology,研究跨尺度的力学和物理性质,以及它们与其他调节机制的相互作用[22];生物符号学,分析有机体或其他生物系统的符号关系系统;生理组学,生物学中生理的系统研究。
癌症系统生物学 Cancer systems biology是系统生物学研究的一个例子,它可以通过特定的研究对象(肿瘤发生和癌症治疗)来区分。它使用特定的数据(患者样本、高通量数据,特别注意在患者肿瘤样本中描述癌症基因组)和工具(永生化癌细胞系、肿瘤发生的小鼠模型、异种移植模型、高通量测序方法、基于siRNA的基因敲除高通量筛选、体细胞突变后果的计算模型和基因不稳定性)。[23]癌症系统生物学的长期目标是能够更好地诊断癌症,对癌症进行分类,并更好地预测建议的治疗结果,这是个性化癌症医学和虚拟癌症患者在更远的前景的基础。在癌症的计算系统生物学方面已经做出了重大的努力,在各种肿瘤的计算机模型中创造了真实的多尺度。
这些研究经常与大规模的微扰方法结合,包括基于基因的(RNA干扰,野生型和突变型基因的错误表达)和使用小分子库的化学方法。机器人和自动化传感器使这种大规模的实验和数据采集成为可能。这些技术仍在出现,并且很多面临产生的数据量越大,质量就越低的问题。各种各样的定量科学家(计算生物学家、统计学家、数学家、计算机科学家和物理学家)正在努力提高这些方法的质量,并创建、完善和重新测试模型,以准确地反映观测结果。
系统生物学方法经常涉及机制模型的发展,比如从动态系统的基本构件的定量特性重建动态系统。例如,一个细胞网络可以进行数学建模,使用的方法来自化学动力学和控制理论。由于细胞网络中参数、变量和约束的数量庞大,系统生物学经常使用数值和如流平衡分析的计算技术。[24]
生物信息学和数据分析
计算机科学、信息学和统计学的其他方面也用于系统生物学。包括新形式的计算模型,如使用过程计算模拟生物过程(著名的方法包括随机演算,BioAmbients,Beta Binders,BioPEPA 和 Brane 演算)和基于约束的建模; 使用信息提取和文本挖掘技术,综合来自文献的信息;[25]开发在线数据库和存储库共享数据和模型,以及通过软件,网站和数据库或商业诉讼的松散耦合实现数据库集成和软件互操作性的方法;基于网络的方法分析高维基因组数据集。例如,加权相关网络分析常常用于识别集群(称为模块)、建立集群之间的关系模型、计算集群(模块)成员的模糊度量、识别模块内中心成员,以及利用其他数据集研究集群保存; 基于通路的组学数据分析方法,例如识别和评价不同活性的基因、蛋白质或代谢物通路的方法。[26] 许多基因组数据集的分析也包括确定相关性。此外,由于大量的信息来自不同的领域,发展生物模型的语法和语义健全的表示方法是必要的。[27]
建立生物学模型
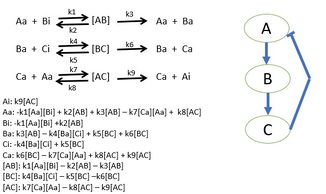
研究人员首先选择一条生物通路,绘制所有蛋白质相互作用的图表。确定了所有的蛋白质相互作用之后,使用符合质量作用定律的动力学来描述系统中反应的速率。质量作用定律动力学将提供微分方程,把生物系统模拟成一个数学模型,其中微分方程的参数可以由实验来确定。[29]这些参数值是系统中每对蛋白质相互作用的反应速率。这个模型决定了生物系统中主要蛋白质的行为,并且为理解单个蛋白质的特殊行为提供了新的视角。当不能够收集一整个系统的所有反应速率时。可以通过模拟已知参数的模型并且提供可能参数值的目标行为,来确定未知的反应速率。[30]
参见
- 生物计算 Biological computation
- 计算生物学 Computational biology
- 暴露组 Exposome
- 相互作用组 Interactome
- 生物学中组学主题列表 List of omics topics in biology
- 代谢网络建模Metabolic network modelling
- 生物系统建模Modelling biological systems
- 分子病理流行病学 Molecular pathological epidemiology
- 网络生物学 Network biology
- 网络医药 Network medicine
- 合成生物学 Synthetic biology
- 系统生物医学 Systems biomedicine
- 系统免疫学 Systems immunology
参考文献
- ↑ 1.0 1.1 Tavassoly, Iman; Goldfarb, Joseph; Iyengar, Ravi (2018-10-04). "Systems biology primer: the basic methods and approaches". Essays in Biochemistry. 62 (4): 487–500. doi:10.1042/EBC20180003. ISSN 0071-1365. PMID 30287586.
- ↑ Bu Z, Callaway DJ (2011). "Proteins MOVE! Protein dynamics and long-range allostery in cell signaling". Protein Structure and Diseases. Advances in Protein Chemistry and Structural Biology. 83. pp. 163–221. doi:10.1016/B978-0-12-381262-9.00005-7. ISBN 978-0-123-81262-9. PMID 21570668.
- ↑ Voit, Eberhard (2012). A First Course in Systems Biology. Garland Science. ISBN 9780815344674.
- ↑ Baitaluk, M. (2009). "System Biology of Gene Regulation". Biomedical Informatics. Methods in Molecular Biology. 569. pp. 55–87. doi:10.1007/978-1-59745-524-4_4. ISBN 978-1-934115-63-3. PMID 19623486.
- ↑ Turing, A. M. (1952). "The Chemical Basis of Morphogenesis" (PDF). Philosophical Transactions of the Royal Society B: Biological Sciences. 237 (641): 37–72. Bibcode:1952RSPTB.237...37T. doi:10.1098/rstb.1952.0012. JSTOR 92463.
- ↑ Zou, Yawen; Laubichler, Manfred D. (2018-07-25). "From systems to biology: A computational analysis of the research articles on systems biology from 1992 to 2013". PLOS ONE (in English). 13 (7): e0200929. Bibcode:2018PLoSO..1300929Z. doi:10.1371/journal.pone.0200929. ISSN 1932-6203. PMC 6059489. PMID 30044828.
- ↑ B. J. Zeng, "On the holographic model of human body", 1st National Conference of Comparative Studies Traditional Chinese Medicine and West Medicine, Medicine and Philosophy, April 1992 ("systems medicine and pharmacology" termed).
- ↑ Zeng (B.) J., On the concept of system biological engineering, Communication on Transgenic Animals, No. 6, June, 1994.
- ↑ B. J. Zeng, "Transgenic animal expression system – transgenic egg plan (goldegg plan)", Communication on Transgenic Animal, Vol.1, No.11, 1994 (on the concept of system genetics and term coined).
- ↑ B. J. Zeng, "From positive to synthetic science", Communication on Transgenic Animals, No. 11, 1995 (on systems medicine).
- ↑ B. J. Zeng, "The structure theory of self-organization systems", Communication on Transgenic Animals, No.8-10, 1996. Etc.
- ↑ B. J. Zeng, "The structure theory of self-organization systems", Communication on Transgenic Animals, No.8-10, 1996. Etc.
- ↑ "HMS launches new department to study systems biology". Harvard Gazette. September 23, 2003.
- ↑ Ayyadurai, VA; Dewey, CF (March 2011). "CytoSolve: A Scalable Computational Method for Dynamic Integration of Multiple Molecular Pathway Models". Cell Mol Bioeng. 4 (1): 28–45. doi:10.1007/s12195-010-0143-x. PMC 3032229. PMID 21423324.
- ↑ "Systems Biology - National Institute of General Medical Sciences". Archived from the original on 19 October 2013. Retrieved 12 December 2012.
- ↑ Karr, Jonathan R.; Sanghvi, Jayodita C.; Macklin, Derek N.; Gutschow, Miriam V.; Jacobs, Jared M.; Bolival, Benjamin; Assad-Garcia, Nacyra; Glass, John I.; Covert, Markus W. (July 2012). "A Whole-Cell Computational Model Predicts Phenotype from Genotype". Cell. 150 (2): 389–401. doi:10.1016/j.cell.2012.05.044. PMC 3413483. PMID 22817898.
- ↑ Cascante, Marta; Marin, Silvia (2008-09-30). "Metabolomics and fluxomics approaches". Essays in Biochemistry (in English). 45: 67–82. doi:10.1042/bse0450067. ISSN 0071-1365. PMID 18793124.
- ↑ Cusick, Michael E.; Klitgord, Niels; Vidal, Marc; Hill, David E. (2005-10-15). "Interactome: gateway into systems biology". Human Molecular Genetics (in English). 14 (suppl_2): R171–R181. doi:10.1093/hmg/ddi335. ISSN 0964-6906. PMID 16162640.
- ↑ Aur, Dorian (2012). "From Neuroelectrodynamics to Thinking Machines". Cognitive Computation (in English). 4 (1): 4–12. doi:10.1007/s12559-011-9106-3. ISSN 1866-9956.
- ↑ Diez, Mikel; Petuya, Víctor; Martínez-Cruz, Luis Alfonso; Hernández, Alfonso (2011-12-01). "A biokinematic approach for the co--mputational simulation of proteins molecular mechanism". Mechanism and Machine Theory. 46 (12): 1854–1868. doi:10.1016/j.mechmachtheory.2011.07.013. ISSN 0094-114X.
- ↑ Loor, Khuram Shahzad and Juan J. (2012-07-31). "Application of Top-Down and Bottom-up Systems Approaches in Ruminant Physiology and Metabolism". Current Genomics (in English). 13 (5): 379–394. doi:10.2174/138920212801619269. PMC 3401895. PMID 23372424.
- ↑ Spill, Fabian; Bakal, Chris; Mak, Michael (2018). "Mechanical and Systems Biology of Cancer". Computational and Structural Biotechnology Journal. 16: 237–245. arXiv:1807.08990. Bibcode:2018arXiv180708990S. doi:10.1016/j.csbj.2018.07.002. PMC 6077126. PMID 30105089.
- ↑ Barillot, Emmanuel; Calzone, Laurence; Hupe, Philippe; Vert, Jean-Philippe; Zinovyev, Andrei (2012). Computational Systems Biology of Cancer. Chapman & Hall/CRCMathematical & Computational Biology. p. 461. ISBN 978-1439831441.
- ↑ Tavassoly, Iman (2015). Dynamics of Cell Fate Decision Mediated by the Interplay of Autophagy and Apoptosis in Cancer Cells. Springer Theses. Springer International Publishing. doi:10.1007/978-3-319-14962-2. ISBN 978-3-319-14961-5.
- ↑ Ananadou, Sophia; Kell, Douglas; Tsujii, Jun-ichi (December 2006). "Text mining and its potential applications in systems biology". Trends in Biotechnology. 24 (12): 571–579. doi:10.1016/j.tibtech.2006.10.002. PMID 17045684.
- ↑ Glaab, Enrico; Schneider, Reinhard (2012). "PathVar: analysis of gene and protein expression variance in cellular pathways using microarray data". Bioinformatics. 28 (3): 446–447. doi:10.1093/bioinformatics/btr656. PMC 3268235. PMID 22123829.
- ↑ Bardini, R.; Politano, G.; Benso, A.; Di Carlo, S. (2017-01-01). "Multi-level and hybrid modelling approaches for systems biology". Computational and Structural Biotechnology Journal. 15: 396–402. doi:10.1016/j.csbj.2017.07.005. ISSN 2001-0370. PMC 5565741. PMID 28855977.
- ↑ Transtrum, Mark K.; Qiu, Peng (2016-05-17). "Bridging Mechanistic and Phenomenological Models of Complex Biological Systems". PLOS Computational Biology. 12 (5): e1004915. arXiv:1509.06278. Bibcode:2016PLSCB..12E4915T. doi:10.1371/journal.pcbi.1004915. ISSN 1553-7358. PMC 4871498. PMID 27187545.
- ↑ Chellaboina, V.; Bhat, S. P.; Haddad, W. M.; Bernstein, D. S. (August 2009). "Modeling and analysis of mass-action kinetics". IEEE Control Systems Magazine. 29 (4): 60–78. doi:10.1109/MCS.2009.932926. ISSN 1941-000X.
- ↑ Brown, Kevin S.; Sethna, James P. (2003-08-12). "Statistical mechanical approaches to models with many poorly known parameters". Physical Review E. 68 (2): 021904. Bibcode:2003PhRvE..68b1904B. doi:10.1103/physreve.68.021904. ISSN 1063-651X. PMID 14525003.
延伸阅读
- Klipp, Edda; Liebermeister, Wolfram; Wierling, Christoph; Kowald, Axel (2016). Systems Biology - A Textbook, 2nd edition. Wiley. ISBN 978-3-527-33636-4.
- Asfar S. Azmi, ed. (2012). Systems Biology in Cancer Research and Drug Discovery. ISBN 978-94-007-4819-4.
- Kitano, Hiroaki (15 October 2001). Foundations of Systems Biology. MIT Press. ISBN 978-0-262-11266-6. https://archive.org/details/foundationsofsys00hiro.
- Werner, Eric (29 March 2007). "All systems go". Nature. 446 (7135): 493–494. Bibcode:2007Natur.446..493W. doi:10.1038/446493a. provides a comparative review of three books:
- Alon, Uri (7 July 2006). An Introduction to Systems Biology: Design Principles of Biological Circuits. Chapman & Hall. ISBN 978-1-58488-642-6.
- Kaneko, Kunihiko (15 September 2006). Life: An Introduction to Complex Systems Biology. Springer-Verlag. Bibcode 2006lics.book.....K. ISBN 978-3-540-32666-3.
- Palsson, Bernhard O. (16 January 2006). Systems Biology: Properties of Reconstructed Networks. Cambridge University Press. ISBN 978-0-521-85903-5.
- Encyclopedia of Systems Biology. Springer-Verlag. 13 August 2013. ISBN 978-1-4419-9864-4.
相关链接
编者推荐
集智文章推荐
生命是怎样涌现的:系统生物学入门全路径
- 系统生物学及相关跨学科领域正在兴起,近年来基于各类疾病的组学研究成果频出。作者整理了这份结合多门教材、多篇经典论文的学习路径,供你入门参考。
意识如何自下而上地涌现?来自系统生物学的启示 | 长文综述
- 智识健全的成年人通过内省而认知、感受到的“意识”,被称为现象意识。这往往是与意识相关的定义中最让人摸不着头脑、而又最令人着迷的概念。然而它终究是从一些不那么复杂的事物中,自下而上地涌现出来的。但即使是在研究意识的科学社群,也很少有人深入思考这一点
集智课程推荐
系统科学导引(一):概论部分
- 本课程以实际研究工作的例子来阐述什么是系统科学,内容丰富,可以帮助学习者把数学物理学的概念思想分析方法用于具有系统科学特点的实际问题。
Scaling in Biology and Society
- 本课程中,讲解在生物系统和社会系统中的规模特性。
本中文词条由CecileLi、Thingamabob审校,许许编辑,欢迎在讨论页面留言。
本词条内容源自wikipedia及公开资料,遵守 CC3.0协议。